sim1 = pd.read_csv("data/sim1.csv")Model Basics
R for Data Science by Wickham & Grolemund
Introduction
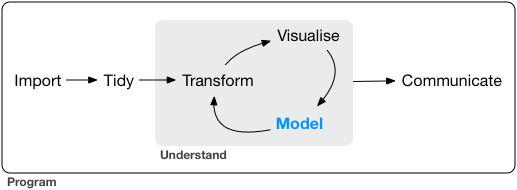
Source: R for Data Science by Wickham & Grolemund
모델의 목표는 데이터 세트에 대한 간단한 저차원 요약을 제공하는 것입니다. 이상적으로, 모델은 진정한 ‘신호’(즉, 관심 있는 현상에 의해 생성된 패턴)를 포착하고 ‘노이즈’(즉, 관심 없는 임의의 변동)는 무시합니다. (번역 by DeepL)
The goal of a model is to provide a simple low-dimensional summary of a dataset. Ideally, the model will capture true “signals” (i.e. patterns generated by the phenomenon of interest), and ignore “noise” (i.e. random variation that you’re not interested in).
이상적으로, 모형(model)이 현상으로부터 노이즈가 제거된 진정한 신호를 잡아내 주기를 기대.
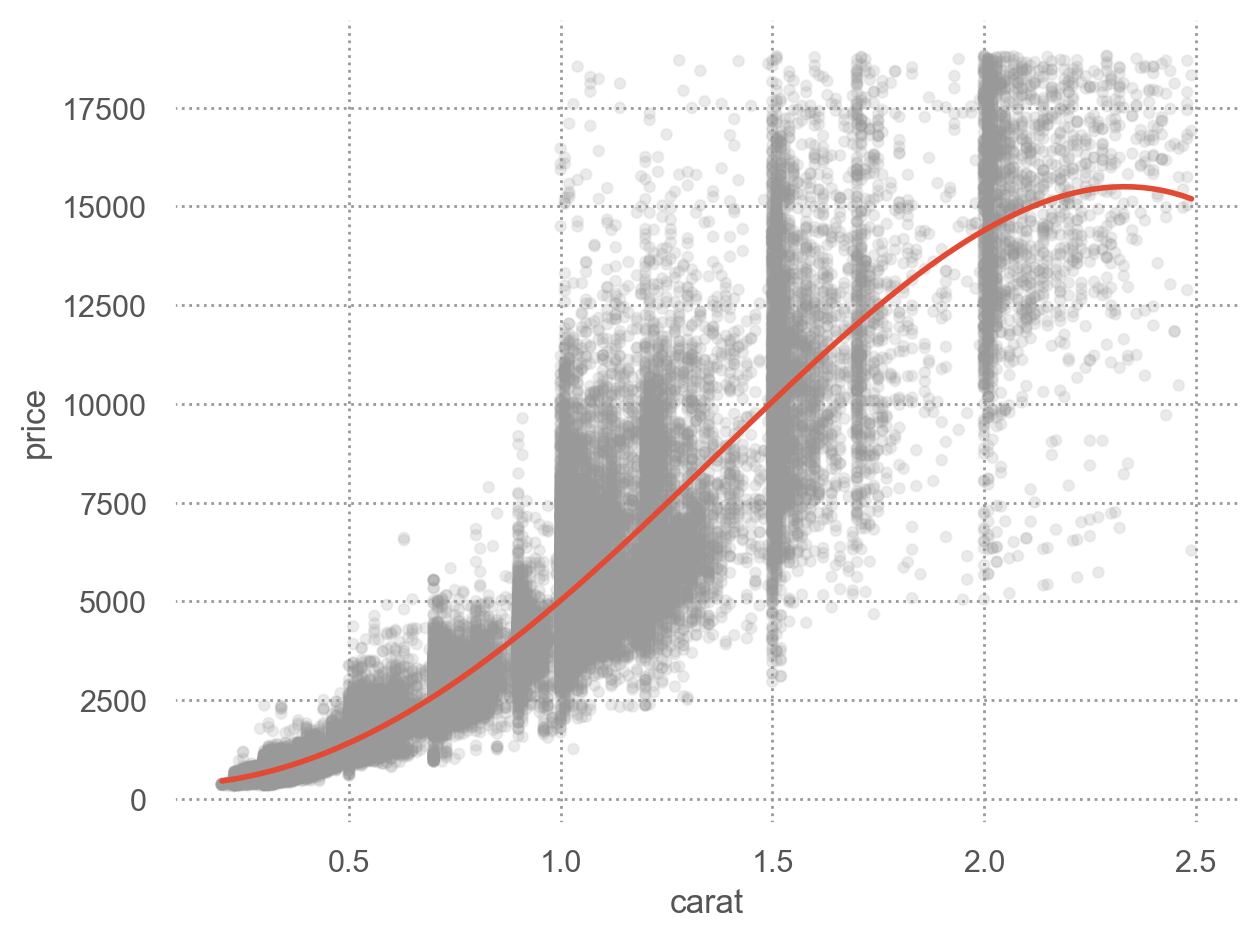
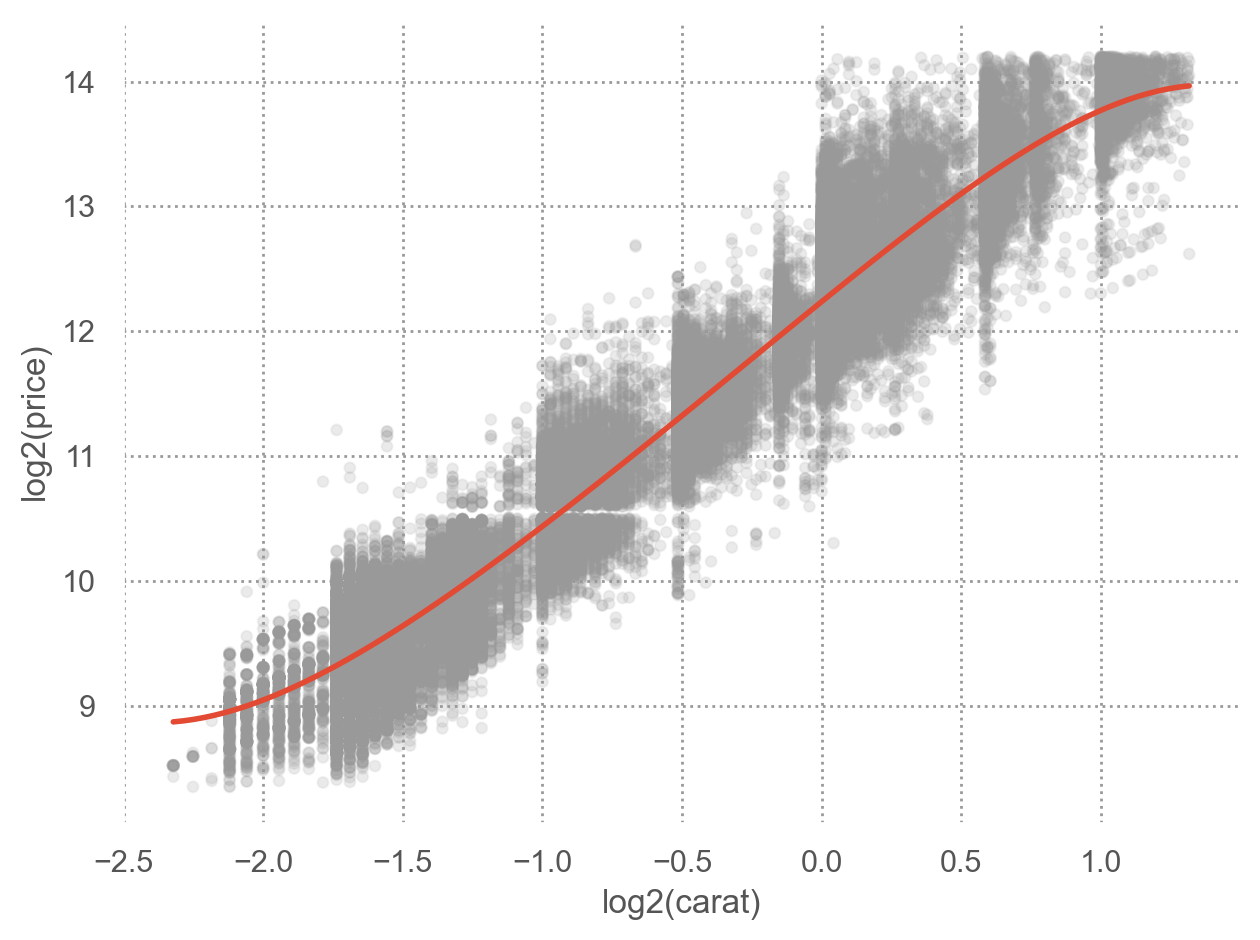
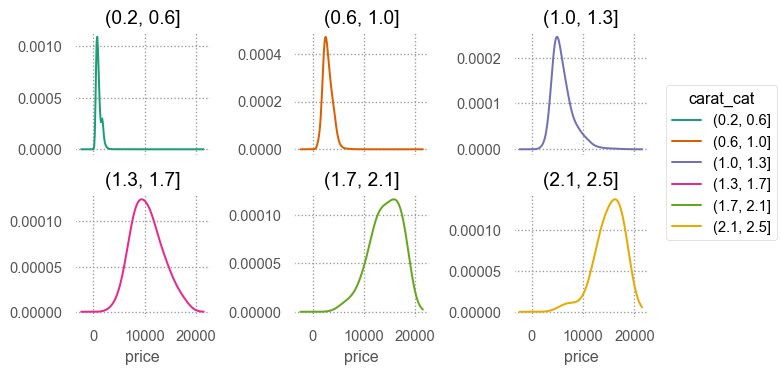
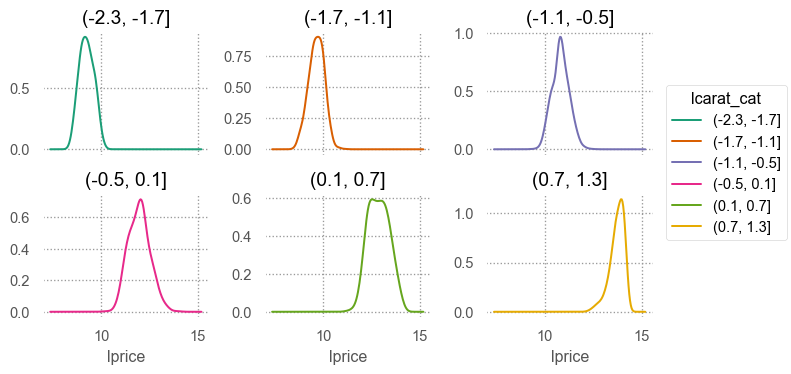
예를 들어, 캐럿과 가격의 진정한 관계를 모델로 표현
Linear model: \(Y = aX + b + \epsilon\), \(\epsilon\): errors
- \(price = a \cdot carat + b + \epsilon\)
- 로그 변환 후 선형관계로 가정: \(log(price) = a \cdot log(carat) + b + \epsilon\)
- \(\epsilon\): Gaussian 분포로 가정, 즉 \(\epsilon \sim N(0, \sigma^2)\)
- \(E(Y|X = X_i) = a \cdot X_i + b\) (\(E\): expectation, 기대값)
- 즉, \(X = X_i\)에 대한 conditional mean이 선형함수로 결정됨을 가정
Errors은 노이즈?
- Reducible error: 모형이 잡아내지 못한 신호; 영향을 미치지만 측정하지 않은 변수가 존재
- Irreducible error:
- 측정 오차 (measurement error): ex. 성별, 젠더, 키, 온도, 강수량, 지능, 불쾌지수, …
- Random processes
- 물리적 세계의 불확실성: stochastic vs. deterministic world
- 예를 들어, 동전을 4번 던질 때
- 보통 Gaussian 분포를 이루거나 가정: ex. 측정 오차들, 동전 앞면의 개수들, 키, 몸무게, IQ, …
- 그 외에 자연스럽게 나타난다고 가정할 수 있는 여러 분포들이 있음; Binomial, Poisson, Exponential, Weibull, Gamma, Beta, …
불확실성(uncertainty)
- 예측은 정확할 수 없으며, 이 불확실성이 error로 표현되며,
- 확률의 개념으로 모형의 일부로 포함되어 예측하는데 중요한 요소로 활용됨.
Gaussian/Normal distribution
- 랜덤한 값들의 합/평균들이 나타내는 분포; 중심극한정리(Central Limit Theorem)
- 측정 오차의 분포(error distribution)
- 다양한 힘들의 상호작용으로 인한 분포
- 분산이 유한한 분포 중에 정보 엔트로피가 최대(maximum entropy)인 분포
- \(X \sim N(\mu, \sigma^2)\); density function \(\displaystyle f(x) = \frac{1}{\sqrt{2\pi}\sigma} e^{-\frac{1}{2}(\frac{x-\mu}{\sigma})^2}\)
- \(X \sim N(0, 1)\); \(\displaystyle f(x) = \frac{1}{\sqrt{2\pi}} e^{-\frac{1}{2}x^2}\), (standard normal distribution)
1000 people, each 16 steps tossing a coin:
[[-1 1 -1 ... -1 1 -1]
[ 1 -1 -1 ... -1 1 1]
[-1 1 -1 ... -1 1 -1]
...
[-1 1 -1 ... -1 1 -1]
[-1 1 -1 ... -1 1 -1]
[-1 -1 1 ... -1 -1 -1]]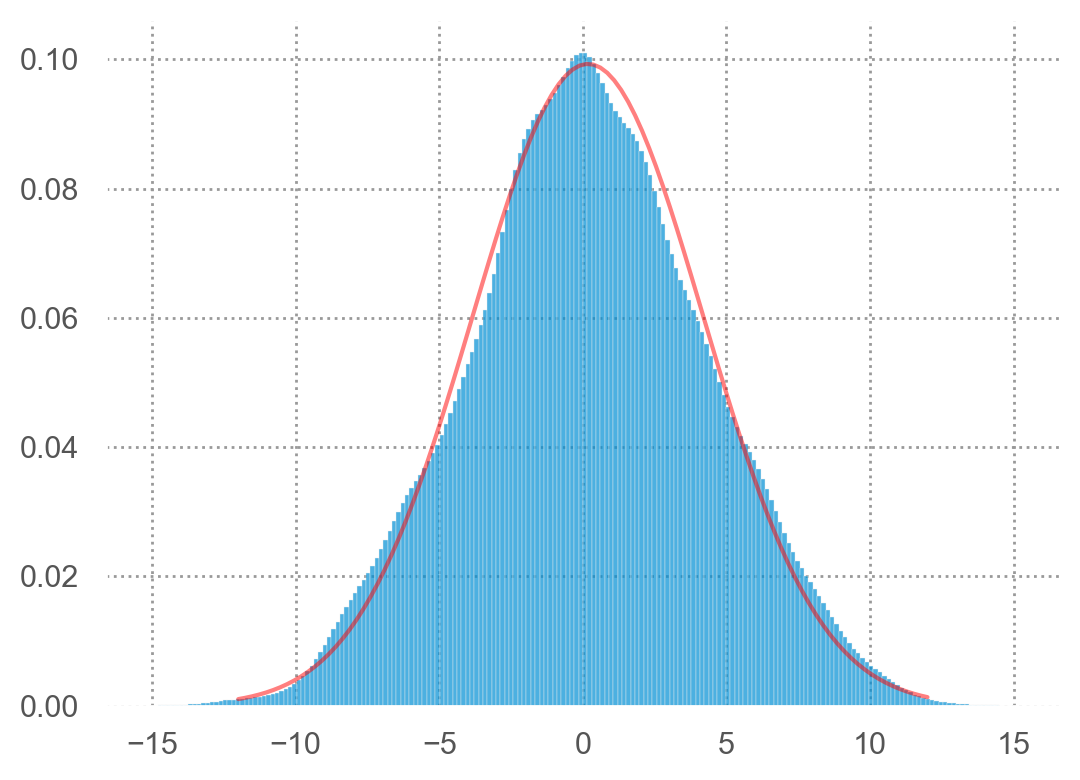
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from scipy.stats import norm
np.random.seed(0)
def plot_Gaussin(n=0, ax=None):
N = 200
num_people, num_rounds = N, n
if n == 0:
toss = np.zeros(N).reshape(N, -1)
else:
toss = np.random.choice([-1, 1], size=(num_people, num_rounds))
toss_sum = toss.sum(axis=1)
ax = ax or plt.gca()
ax.set_title(f"{N} people, each tossing a fair coin {n} times")
df = pd.DataFrame({"TossSum": toss_sum})
df2 = df.value_counts().reset_index()
x = df2["TossSum"].values.astype(int)
y = df2["count"].values
if n < 10:
ax.bar(x, y, color="#1f77b4", alpha=.6, width=.8)
ax.set_xlim(-10, 10)
ax.set_xticks(x)
else:
ax.bar(x, y, color="#1f77b4", alpha=.6)
ax.set_xticks(x)
for i, v in enumerate(y):
ax.text(x[i], v + 0.5, str(v), ha="center", color="r")
from ipywidgets import interact, fixed
interact(plot_Gaussin, n=(0, 100), ax=fixed(None))Model basics
우선, simulated 데이터셋으로 모형을 세우는 방식을 들여다보면서 본질적인 부분을 익히고자 함.
모델은 두 부분으로 나뉘는데
A family of models를 정의: generic 패턴을 표현해 줄 수 있는 모델 클래스
- 선형적인 관계라면 가령, 예측변수가 2개인 선형 모델인 \(y = \beta_0 + \beta_1 x_1 + \beta_2 x_2\)
- 지수적 관계라면 가령, \(y = \beta_0 + \beta_2 x^{\beta_1}\)
\(\beta_0, \beta_1, \beta_2\)는 패턴을 잡아낼 수 있도록 변하는 파라미터(parameter); 통계에서는 모집단(population)에서 변수들의 관계를 의미
머신러닝에서는 parameters, coefficients, weights 등으로 혼용
A fitted model을 생성: 데이터에 가장 가까운(적합한) 파라미터에 해당하는 특정 모델을 선택: “fit a model to data”
- \(y = 3x+7\)
- \(y = 9x^{1.6}\)
A fitted model은 a family of models 중에 데이터에 가장 가까운 모델임
- 이는 소위 “best” model일 뿐
- “good” model임을 뜻하지 않고, “true” model임을 뜻하는 것은 더더욱 아님
All models are wrong, but some are useful.
The goal of a model is not to uncover truth, but to discover a simple approximation that is still useful.
A simple model
Data: sim1.csv
x y
0 1 4.20
1 1 7.51
2 1 2.13
.. .. ...
27 10 24.97
28 10 23.35
29 10 21.98
[30 rows x 2 columns]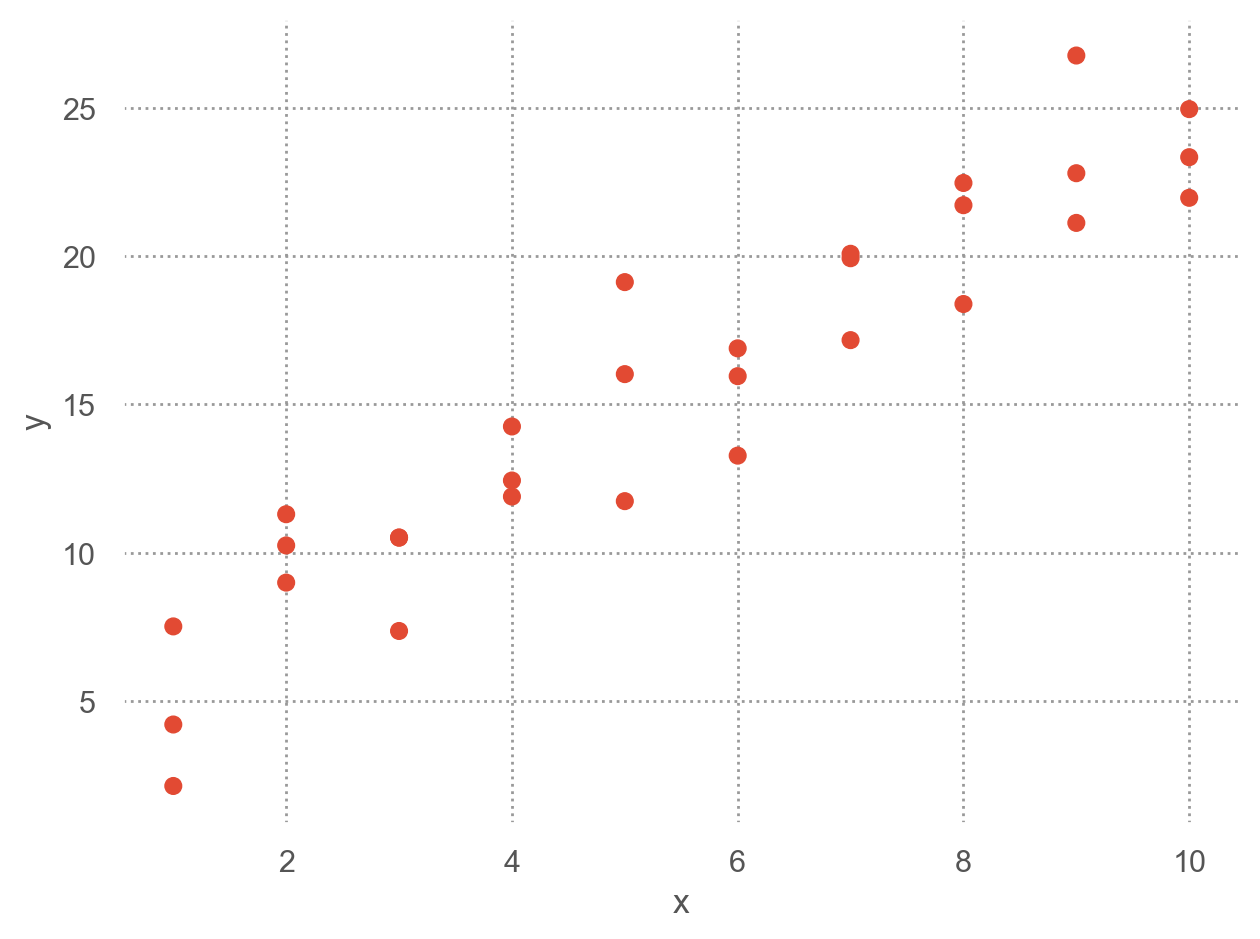
- 패턴: 강한 선형 관계
- 선형 모델 family/class인 \(y = \beta_0 + \beta_1 x\)을 세운 후
- 무수히 많은 \(\beta_0, \beta_1\)의 값들 중 위 데이터에 가장 가까운 값을 찾음
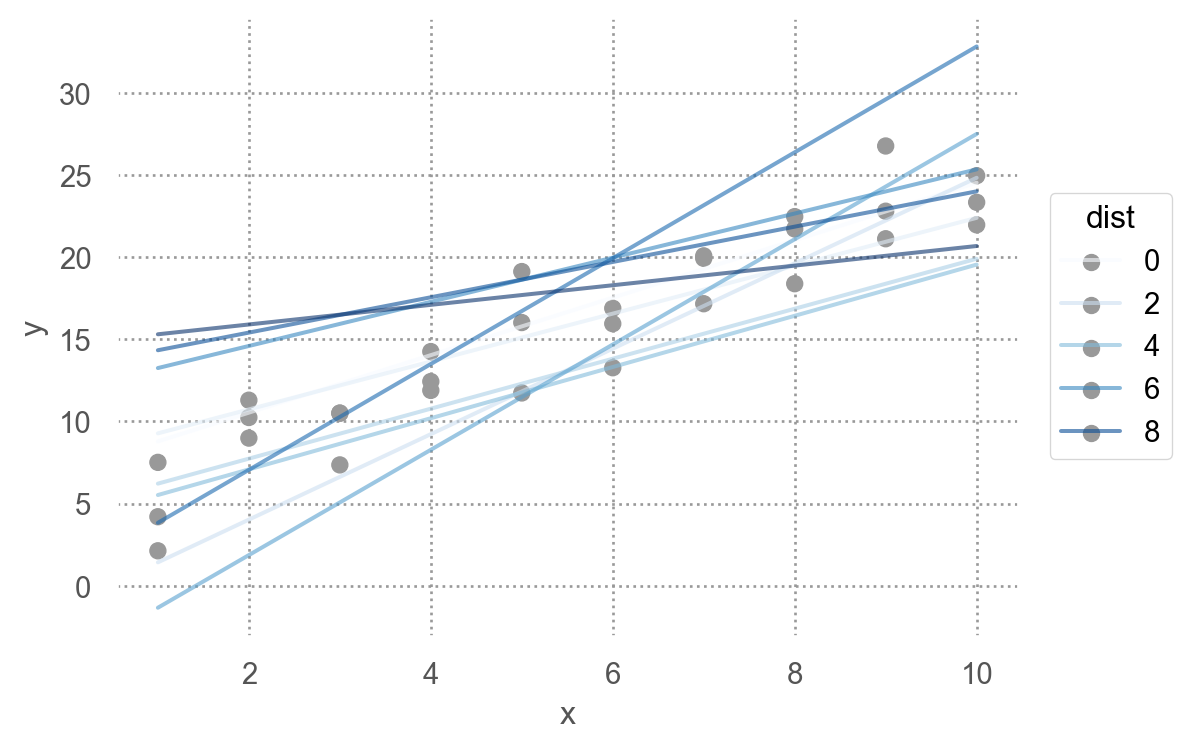
- 그 예로, 임의로 250개의 선형 모델을 그려보면,
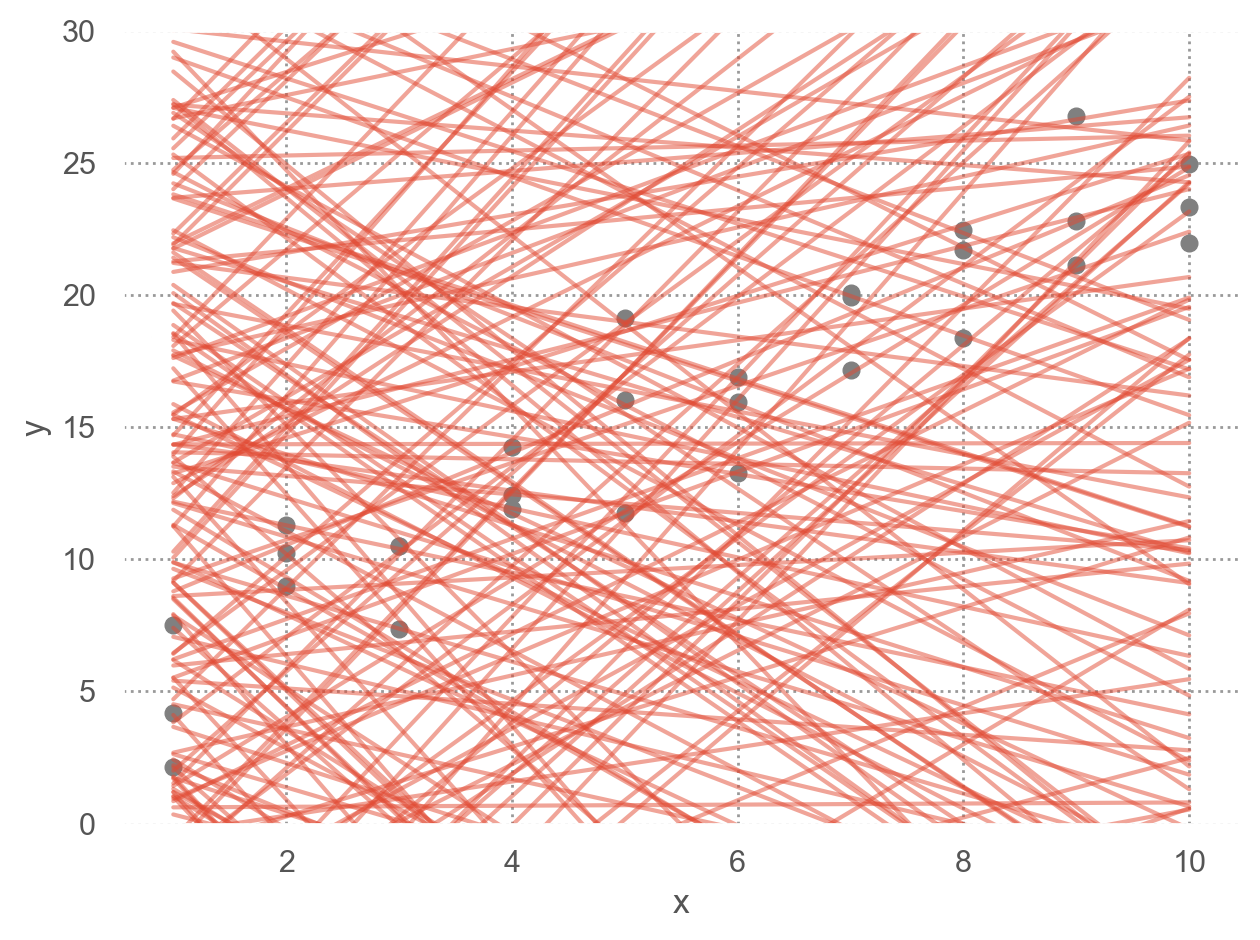
이 선형모델 중 데이터에 가장 가까운 모델을 찾고자 하는데, 이를 위해서는 데이터와 모델과의 거리를 정의해야 함.
\(d =|~data - model~|\)
예) 모델과 데이터의 수직 거리(residuals)의 총체
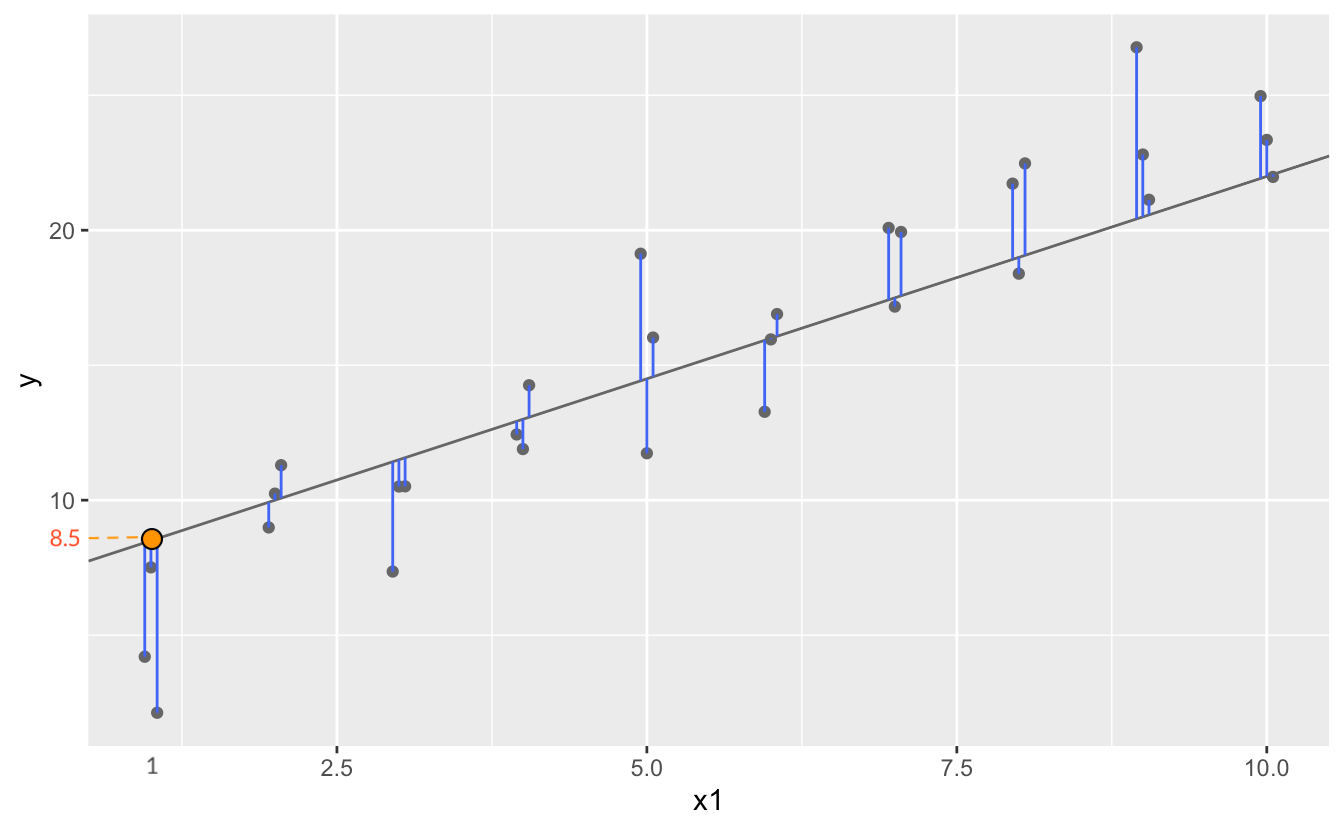
Model 1.1: \(y = 1.5x+7\)의 경우, 이 모델이 예측하는 값들
array([ 8.5, 8.5, 8.5, 10. , 10. , 10. , 11.5, 11.5, 11.5, 13. , 13. ,
13. , 14.5, 14.5, 14.5, 16. , 16. , 16. , 17.5, 17.5, 17.5, 19. ,
19. , 19. , 20.5, 20.5, 20.5, 22. , 22. , 22. ])이 때, 관측치(\(Y_i\))와 예측치(\(\hat{Y}_i\))의 차이, \(Y_i - \hat{Y}_i\)를 잔차(residuals) 또는 예측 오차(errors)라고 함
x y pred resid | e
0 1 4.20 8.50 -4.30
1 1 7.51 8.50 -0.99
2 1 2.13 8.50 -6.37
.. .. ... ... ...
27 10 24.97 22.00 2.97
28 10 23.35 22.00 1.35
29 10 21.98 22.00 -0.02
[30 rows x 4 columns]RMSE = \(\displaystyle\sqrt{\frac{1}{n} \sum_{i=1}^{n}{e^2}}\) = 2.67
MAE = \(\displaystyle\frac{1}{n} \sum_{i=1}^{n}|~e~|\) = 1.43
Error functions
- Root-mean-squared error: \(RMSE = \displaystyle\sqrt{\frac{1}{n} \sum_{i=1}^{n}{(y_i -\hat y_i)^2}}\)
- Mean absolute error: \(MAE = \displaystyle\frac{1}{n} \sum_{i=1}^{n}{|~y_i -\hat y_i~|}\) : 극단값들에 덜 민감함
즉, 데이터셋 sim1과 model 1.1 과의 거리를 RMSE로 정의하면, \(d=|~sim1 -model1~| = 2.67\)
위의 250개의 모델에 대해 각각 거리를 구하면
b0 b1 dist
0 21.79 -2.92 17.42
1 -2.83 -0.57 22.83
2 -6.39 2.16 10.26
.. ... ... ...
247 0.51 4.19 10.38
248 27.94 -0.84 11.59
249 27.93 2.45 25.99
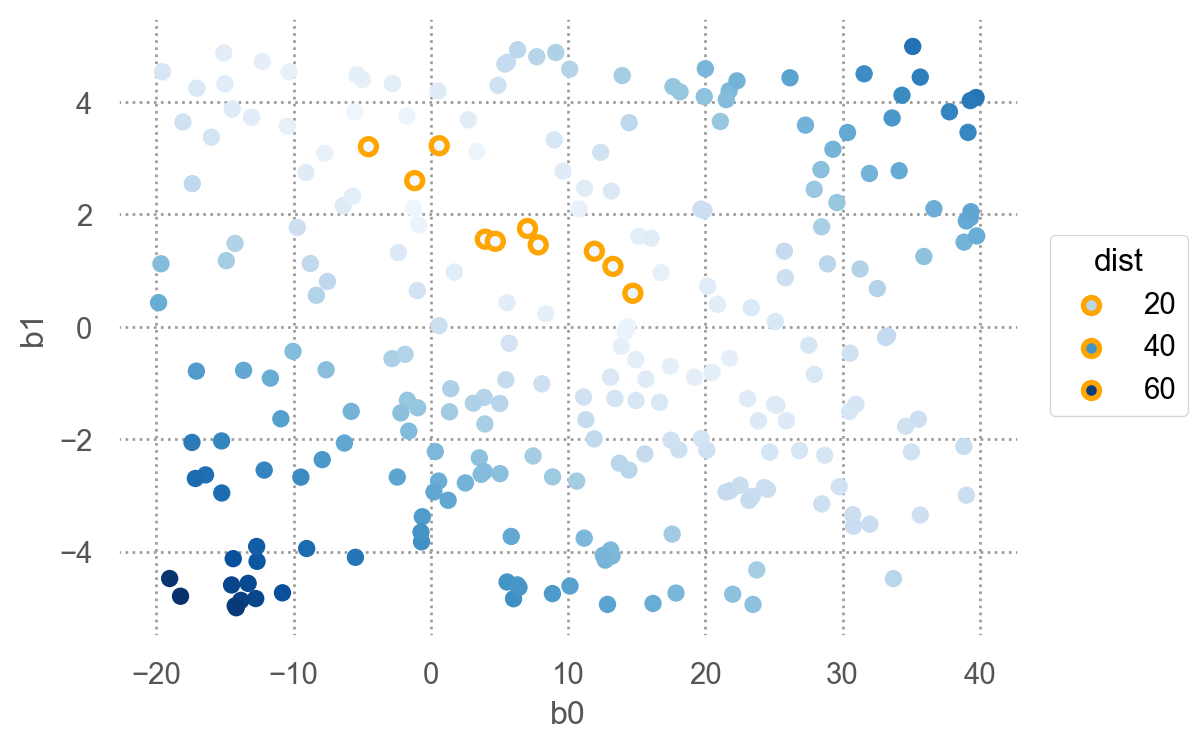
[250 rows x 3 columns]이 중 제일 좋은 모델(dist가 최소) 10개의 모델을 그리면,
250개의 모델 중 10개의 모델을 다음과 같은 \((\beta_0, \beta_1)\) 평면으로 살펴보면, 즉, model space에서 살펴보면
- 오렌지 색은 위에서 구한 10 best models
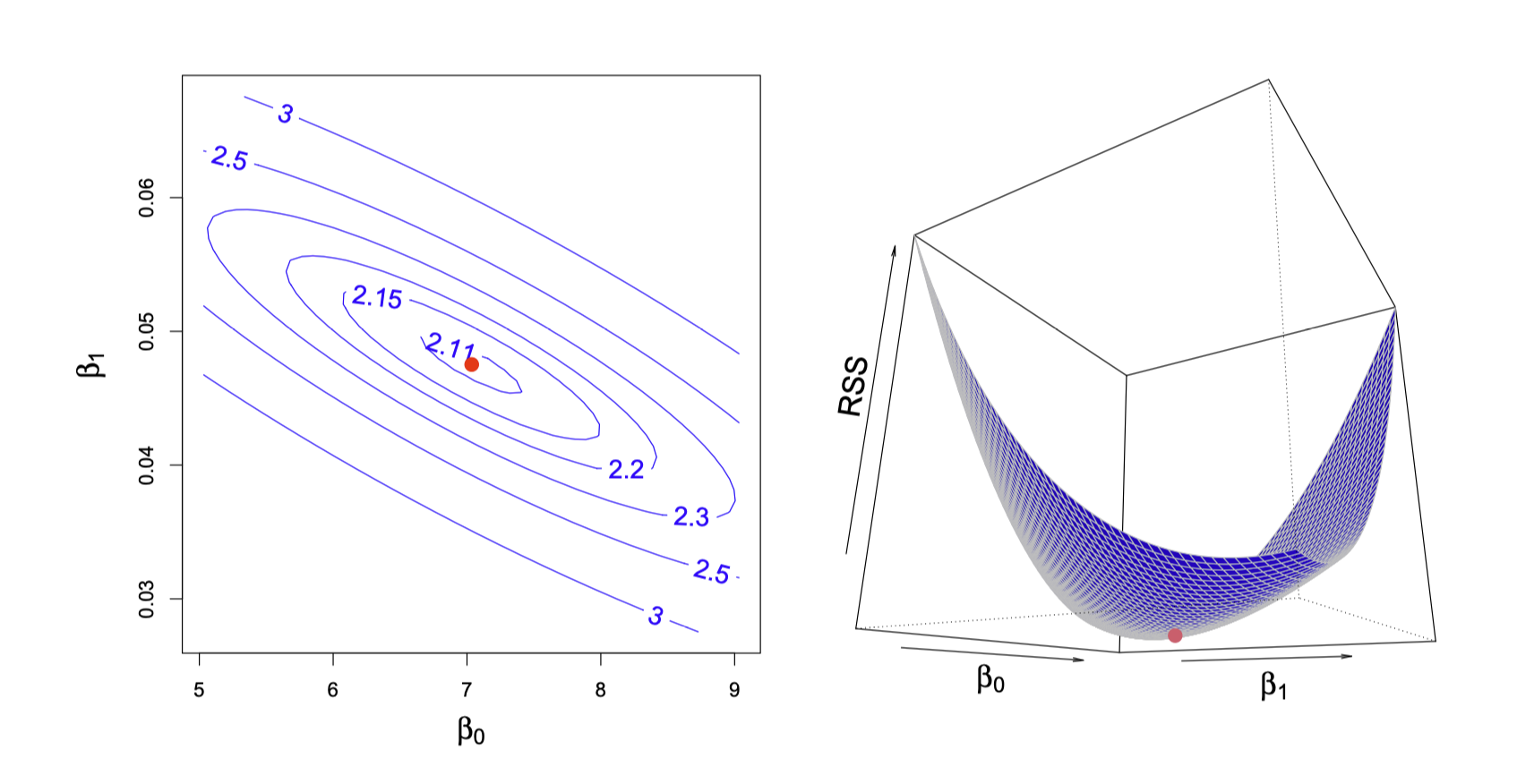
Source: Introduction to Statistical Learning by James et al.
점차 촘촘한 간격으로 grid search를 하면서 거리를 최소로 하는 모델을 찾아가는 것이고, 실제로는 Newton-Raphson search를 통해 최소값을 구하는 알고리즘을 통해 구할 수 있음.
즉, 거리를 최소로 하는 \(\beta_0\), \(\beta_1\)를 찾으면,
from scipy.optimize import minimize
minimize(measure_distance, [0, 0], args=(sim1)).xarray([4.22, 2.05])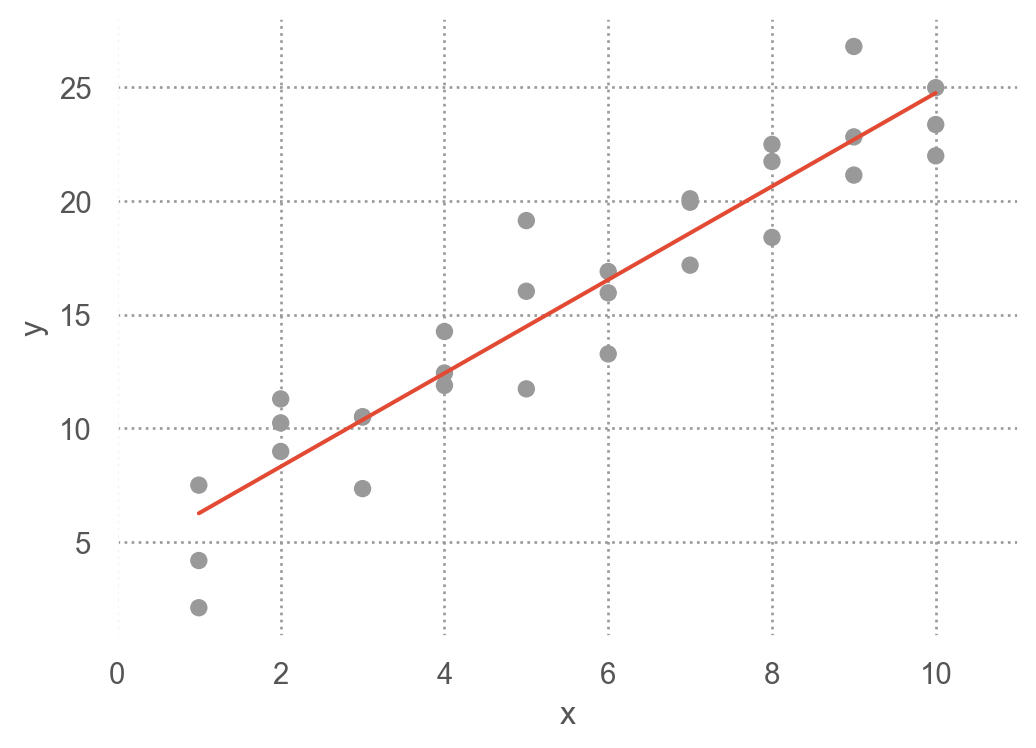
이렇게 squared error가 최소가 되도록 추정하는 것을 ordinary least squares(OLS) estimattion라고 함.
실제로는 위에서 처럼 grid search를 하지 않고, closed-form solution을 통해 바로 구할 수 있음.
from statsmodels.formula.api import ols
mod = ols('y ~ x', data=sim1).fit()
mod.summary()| Dep. Variable: | y | R-squared: | 0.885 |
| Model: | OLS | Adj. R-squared: | 0.880 |
| Method: | Least Squares | F-statistic: | 214.7 |
| Date: | Wed, 14 May 2025 | Prob (F-statistic): | 1.17e-14 |
| Time: | 11:42:02 | Log-Likelihood: | -65.226 |
| No. Observations: | 30 | AIC: | 134.5 |
| Df Residuals: | 28 | BIC: | 137.3 |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
| Intercept | 4.2208 | 0.869 | 4.858 | 0.000 | 2.441 | 6.001 |
| x | 2.0515 | 0.140 | 14.651 | 0.000 | 1.765 | 2.338 |
| Omnibus: | 0.125 | Durbin-Watson: | 2.254 |
| Prob(Omnibus): | 0.939 | Jarque-Bera (JB): | 0.333 |
| Skew: | 0.081 | Prob(JB): | 0.847 |
| Kurtosis: | 2.510 | Cond. No. | 13.7 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
다음과 같은 전형적인 Gaussian이 아닌 분포에 대해서는 OLS estimation은 정확하지 않을 수 있음.
Non-constant variance(왼쪽), Poisson distribution(오른쪽)
Predictive Accuracy
전통적인 모형에서는
- 샘플에 대해서 계산된 값은 실제보다 overestimate 되는 경향이 있으므로,
- 이를 보정하는 방식으로 계산: adjusted, shrunken
현대적인 방식으로는
- 샘플을 training/test set으로 나누어서, test set에 대한 예측값을 계산하고, 이를 통해 예측의 정확성을 평가함
- 비슷하게, resampling 방식의 bootstrapping을 통해 해결
주로 사용되는 지표들
- \(RMSE = \displaystyle\sqrt{{\frac{{1}}{{n}} \sum_{{i=1}}^{{n}}{{e^2}}}}\)
- \(MAE = \displaystyle\frac{{1}}{{n}} \sum_{{i=1}}^{{n}}|~e~|\)
- \(R^2 = 1 - \displaystyle\frac{\frac{1}{n} \sum_{i=1}^{n}{(e-0)^2}}{\frac{1}{n} \sum_{i=1}^{n}{(Y-\overline{Y})^2}} = 1 - \frac{V(e)}{V(Y)} = \frac{V(\widehat Y)}{V(Y)}\), \(V(Y) = V(\widehat Y) + V(e)\)
전통적으로 X가 Y를 얼마나 잘 “설명”해주는지에 대한 지표로서 \(R^2\)를 사용함
calculate RMSE, MAE, R2
from statsmodels.tools.eval_measures import rmse, meanabs
ypred = mod.predict(sim1)
y = sim1["y"]
print(f"RMSE = {rmse(y, ypred):.2f} \nMAE = {meanabs(y, ypred):.2f} \nR-squared = {mod.rsquared:.2f}")RMSE = 2.13
MAE = 1.71
R-squared = 0.88Summary
sim1을 이용한 위의 예는 모든 모델에 적용될 수 있음
즉, \(y = f(x1, x2,...)\) 파라메트릭 클래스의 a family of models을 구성한 후
모델 파라미터(parameter)의 추정
- 모델과 데이터와의 거리 \(d =|~data - model~|\)를 정의한 후
- 거리 \(d\)가 최소가 되는 파라미터를 구하면, a family of models 중 best model을 만들 수 있음
- 보통 squared error function을 사용함.
- 이 squared error가 최소가 되도록 하는 것을 ordinary least squares(OLS) estimator라고 함
- Error가 Gaussian 일때, 이 OLS는 적절한 parameter를 추정하게 됨.
- 더 넓은 확률적 프레임워크인 likelihood의 관점에서 접근할 수 있음
- 주어진 데이터가 관측될 가능도(likelihood)가 최대가 되는 파라이터와 분포를 찾는 방식
- Error가 Gaussian 일때, OLS와 (거의) 동일한 결과를 얻음
- Gaussian을 넘어 다양한 분포의 데이터에 적절히 대응 가능
파라미터 추정값의 불확실성(uncertainty)을 추정
- 파라이터 추정값의 불확실성
- 예측값의 불확실성
예측의 정확성(accuracy)을 평가
- 예측값과 실제값의 차이를 나타내는 지표들: RMSE, MAE, R-squared, AIC, BIC
- 전통적 접근: 표본에서 얻은 결과를 모집단에 대해 이론적으로 보정; 모집단에서는 정확도가 낮아질 것임.
- 현대적 접근: 데이터를 training/test set으로 나누어서 test set으로 모형을 평가
새로운 클래스(형태)의 a family of models을 구성해서 위를 반복하면, 여러 다른 모델들을 비교할 수 있음
Python implementation
앞서 다룬 선형 모형, linear (regression) model은 일반적인 \(\hat{y} = \beta_0 + \beta_1 x_1 + \beta_2 x_2 + ~... ~ + \beta_n x_n\) 형태를 띄고,
앞의 예는 \(n=1\) 에 해당하며, \(\hat{y} =\beta_0 +\beta_1x_1\)에 대해서
간결하게 Wilkinson-Rodgers notation이라고 부르는 symbolic notation을 사용하여 표현하고, 통계에서 주로 사용됨.
Formula y ~ x는 \(\hat{y} =\beta_0 +\beta_1x\)으로 해석되어 처리됨.
- 모형과 데이터의 거리인 RMSE가 최소가 되도록 하는 ordinary least square (OLS) estimate인 \(\beta_0, \beta_1\)을 구하면, best model을 구할 수 있고,
- 그 fitted line은 \(\hat{y} = 4.22 + 2.05x\)
from statsmodels.formula.api import ols
sim1_mod = ols("y ~ x", data = sim1)
sim1_mod.fit().params # 모델의 parameter 즉, coefficients를 내줌
# Intercept 4.22
# x 2.05 # 위에서 구한 파라미터값과 동일함(참고) Linear models의 경우 위에서 처럼 optimization을 이용하지 않고 방정식의 해를 구하듯 closed form으로 최소값을 구함
\(n=2\) 인 경우, 즉 두 변수 x1, x2로 y를 예측하는 경우,
- Formula
y ~ x1 + x2는 모형 \(\hat{y} = \beta_0 +\beta_1x_1 + \beta_2x_2\) 을 의미
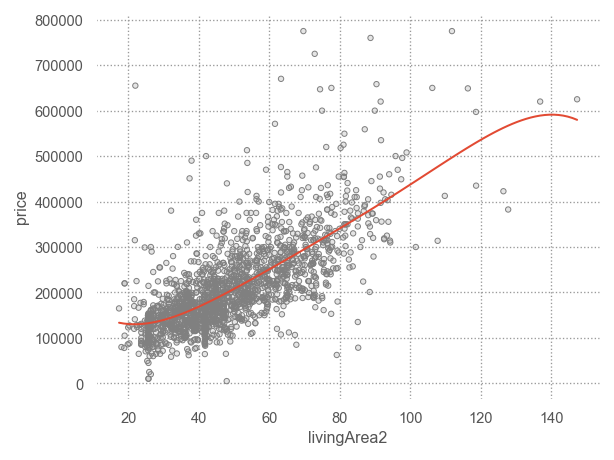
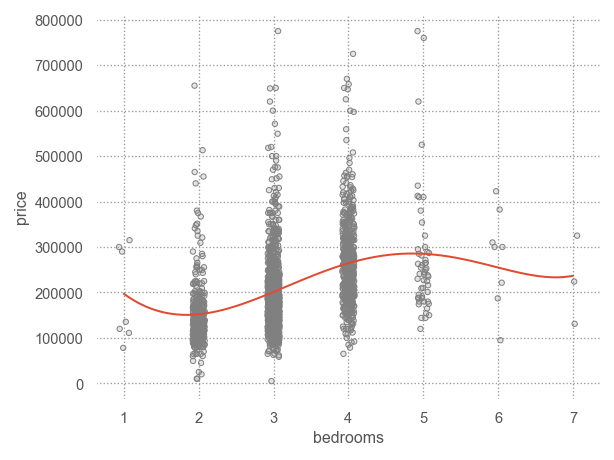
ols("y ~ x1 + x2", data = df)예를 들어, SaratogaHouses 데이터셋에서 집값을 거주공간의 넓이(livingArea)와 침실의 갯수(bedrooms)로 예측하는 경우
houses = sm.datasets.get_rdataset("SaratogaHouses", "mosaicData").data
houses.head(3) price lotSize age landValue livingArea pctCollege bedrooms \
0 132500 0.09 42 50000 906 35 2
1 181115 0.92 0 22300 1953 51 3
2 109000 0.19 133 7300 1944 51 4
fireplaces bathrooms rooms heating fuel sewer \
0 1 1.00 5 electric electric septic
1 0 2.50 6 hot water/steam gas septic
2 1 1.00 8 hot water/steam gas public/commercial
waterfront newConstruction centralAir
0 No No No
1 No No No
2 No No No from statsmodels.formula.api import ols
houses["livingArea2"] = houses["livingArea"] / 35.5
mod_houses = ols("price ~ livingArea2 + bedrooms", data=houses).fit()
mod_houses.paramsIntercept 36667.90
livingArea2 4451.88
bedrooms -14196.77
dtype: float64위와 같이 2개의 예측변수로 평면꼴의 선형모형을 세운다면, 다음과 같은 fitted plane을 구할 수 있음.
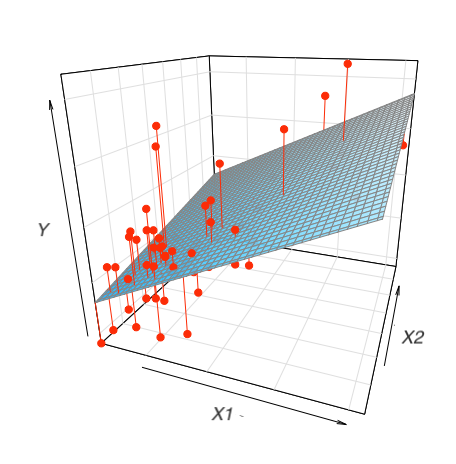
\(\widehat{price} = 36668 + 4452\cdot livingArea2 - 14197 \cdot bedrooms\)
이 계수들의 의미를 파악하는 것이 통계에서 매우 중요한 주제임.
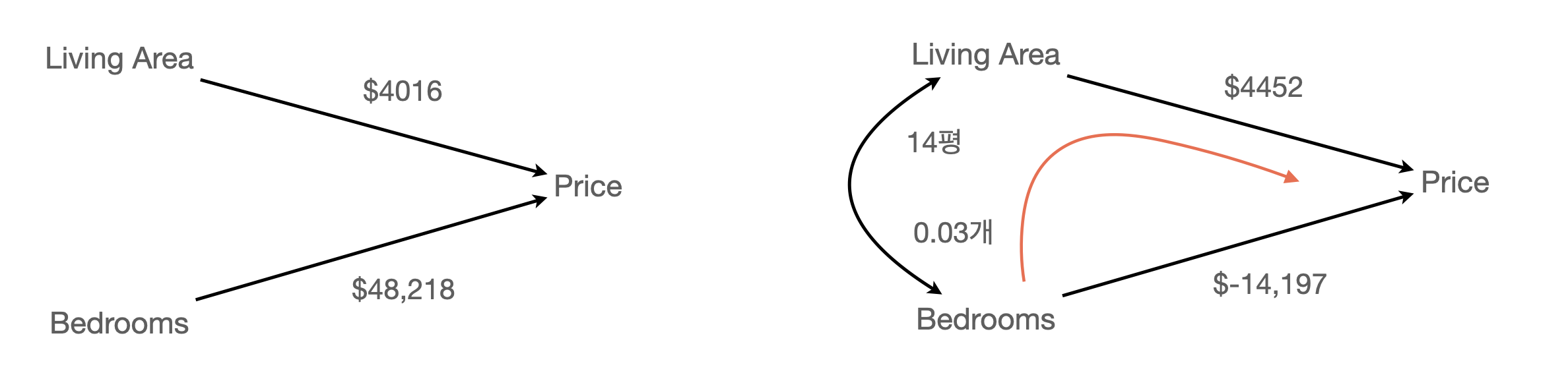
부분 회귀 계수의 해석
다른 변수들이 고정되었을 때, 각 변수의 고유한 효과를 의미함.
같은 의미로, 다른 변수들을 고려(통제, controlling for) 했을 때 혹은 다른 변수의 효과를 제거 (partial out) 했을 때, 각 변수의 고유한 효과를 의미함; holding constant, controlling for, partialing out, adjusted for, residualizing
- 집의 크기와 방의 개수는 깊이 연관되어 있으며, 두 변수의 redunancy가 각 변수들의 효과를 변화시킴.
- 두 예측 변수의 산술적 합으로 연봉을 예측하므로 각 예측변수의 효과는 수정될 수 밖에 없음.
- 수학적으로 보면, 각 예측변수의 기울기는 다른 예측변수의 값에 상관없이 일정하므로, 다른 예측변수들을 (임의의 값에) 고정시키는 효과를 가짐
- 즉, 다른 변수와는 독립적인, 고유한 효과를 추정하게 됨
위의 예에서, 가령 침실의 갯수가 1개 늘면 가격은 $14,197만큼 감소한다는 것은, 집의 크기가 같을 때, 침실의 갯수가 1개 늘어나면 가격이 $14,197만큼 감소한다는 것을 의미함.
- 이는 침실 갯수 자체는 1개 늘면 가격이 $48,218 늘어나는 것으로 보이나
- 실제로 침실 개수가 가격에 미치는 고유한 효과는 -$14,197로 볼 수 있음.
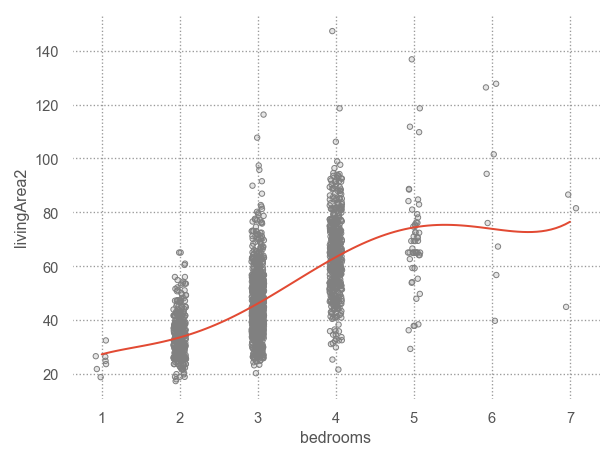
- (빨간색 경로) 침실이 1개 늘면 집의 크기가 14평 늘게 되고 집값은 14평*$4452 = $62,328 늘어는데, 침실 갯수의 효과 $-14,197와 함께, 침실이 1개 늘면 집값은 $62,328 - $14,197 = $48,218 늘어나는 것으로 보였음.
두 변수들 간의 관계를 보면,
mod_houses1 = ols("price ~ livingArea2", data=houses).fit()
mod_houses2 = ols("price ~ bedrooms", data=houses).fit()
mod_houses3 = ols("livingArea2 ~ bedrooms", data=houses).fit()
mod_houses4 = ols("bedrooms ~ livingArea2", data=houses).fit()
display(mod_houses1.params, mod_houses2.params, mod_houses3.params, mod_houses4.params)Intercept 13439.39
livingArea2 4015.85
Intercept 59862.96
bedrooms 48217.81
Intercept 5.21
bedrooms 14.02
Intercept 1.64
livingArea2 0.031
statsmodels는 formula notation으로 모형을 세우는 것을 지원함: statsmodels
sklearn은 formula notation을 직접 지원하지 않지만,patsy패키지를 이용하여 design matrix를 직접 얻어 적용할 수 있음: patsy in scikit-learn 또는 PatsyTransformer
# patsy
from patsy import dmatrices # design matrix
formula = "price ~ livingArea + bedrooms"
y, X = dmatrices(formula, data=houses, return_type="dataframe")
display(y, X) price
0 132500.00
1 181115.00
2 109000.00
... ...
1725 194900.00
1726 125000.00
1727 111300.00
[1728 rows x 1 columns] Intercept livingArea bedrooms
0 1.00 906.00 2.00
1 1.00 1953.00 3.00
2 1.00 1944.00 4.00
... ... ... ...
1725 1.00 1099.00 2.00
1726 1.00 1225.00 3.00
1727 1.00 1959.00 3.00
[1728 rows x 3 columns]# 머신 러닝에서는 보통 직접 입력
y, X = houses["price"], houses[["livingArea", "bedrooms"]]
display(y, X)0 132500
1 181115
2 109000
...
1725 194900
1726 125000
1727 111300
Name: price, Length: 1728, dtype: int64 livingArea bedrooms
0 906 2
1 1953 3
2 1944 4
... ... ...
1725 1099 2
1726 1225 3
1727 1959 3
[1728 rows x 2 columns]Visualising models
Fitted models을 이해하기 위해 모델이 예측하는 부분(prediction)과 모델이 놓친 부분(residuals)을 시각화해서 보는 것이 유용함
Predictions: the pattern that the model has captured
우선, 예측 변수들의 데이터 값을 커버하는 grid를 구성
Data: sim1.csv
sim1 = pd.read_csv("/data/sim1.csv")
sim1 x y
0 1 4.20
1 1 7.51
2 1 2.13
.. .. ...
27 10 24.97
28 10 23.35
29 10 21.98
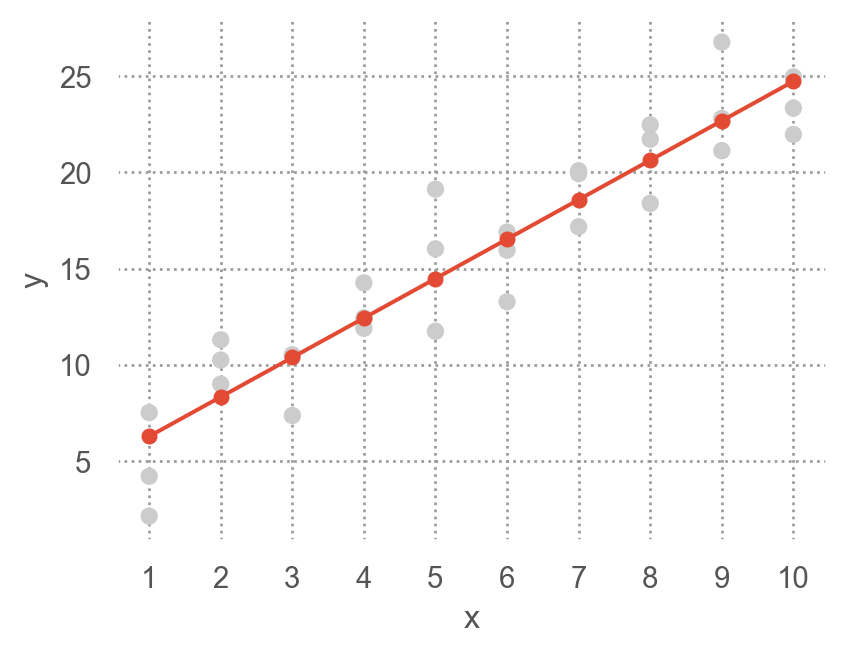
[30 rows x 2 columns]# create a grid for the range of x sim1: new data
grid = pd.DataFrame(dict(x=np.linspace(sim1.x.min(), sim1.x.max(), 10)))모델에 grid를 입력하여 prediction값을 추가
# a model for sim1
from statsmodels.formula.api import ols
sim1_mod = ols("y ~ x", data=sim1).fit()
grid["pred"] = sim1_mod.predict(grid) # column 이름이 매치되어야 함
grid x pred
0 1.00 6.27
1 2.00 8.32
2 3.00 10.38
.. ... ...
7 8.00 20.63
8 9.00 22.68
9 10.00 24.74
[10 rows x 2 columns]prediction을 시각화
Show the code
(
so.Plot(sim1, x='x', y='y')
.add(so.Dot(color=".8"))
.add(so.Line(marker=".", pointsize=10), x=grid.x, y=grid.pred) # prediction!
.layout(size=(4.5, 3.5))
.scale(x=so.Continuous().tick(at=grid.x))
)
Residuals: what the model has missed.
\(e = Y - \hat{Y}\) : 관측값 - 예측값
sim1["fitted"] = sim1_mod.fittedvalues # 또는 sim1_mod.predict(sim1): predicted values (Y_hat)
sim1["resid"] = sim1_mod.resid # Y - Y_hatsim1 x y fitted resid
0 1 4.20 6.27 -2.07
1 1 7.51 6.27 1.24
2 1 2.13 6.27 -4.15
.. .. ... ... ...
27 10 24.97 24.74 0.23
28 10 23.35 24.74 -1.39
29 10 21.98 24.74 -2.76
[30 rows x 4 columns]우선, residuals의 분포를 시각화해서 살펴보면,
sns.set_theme()
sim1["resid"].hist(bins=20);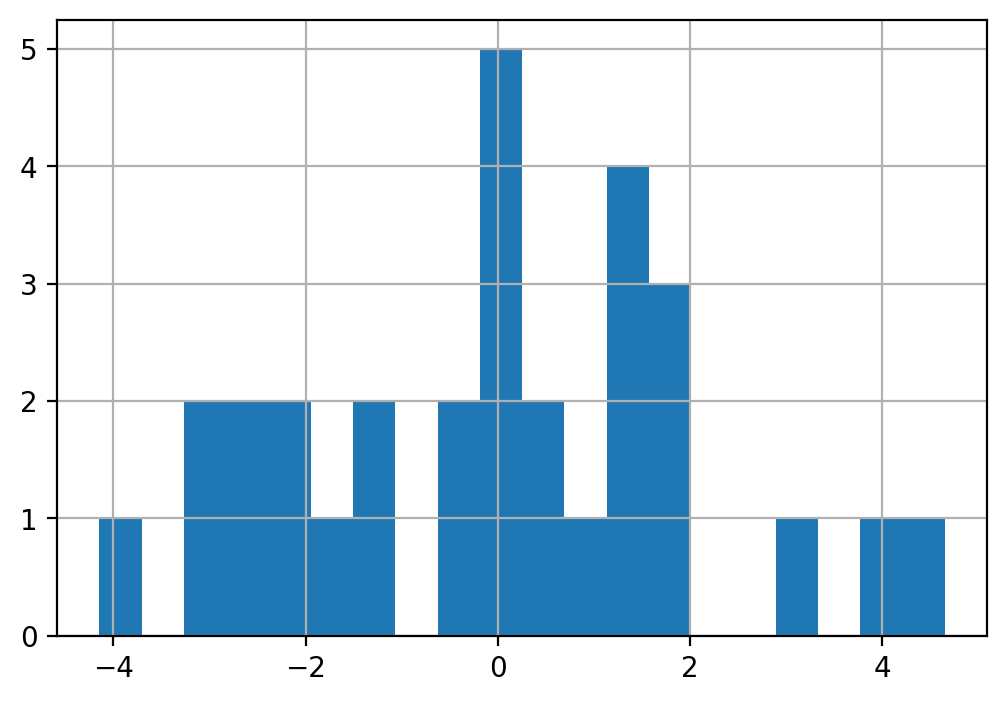
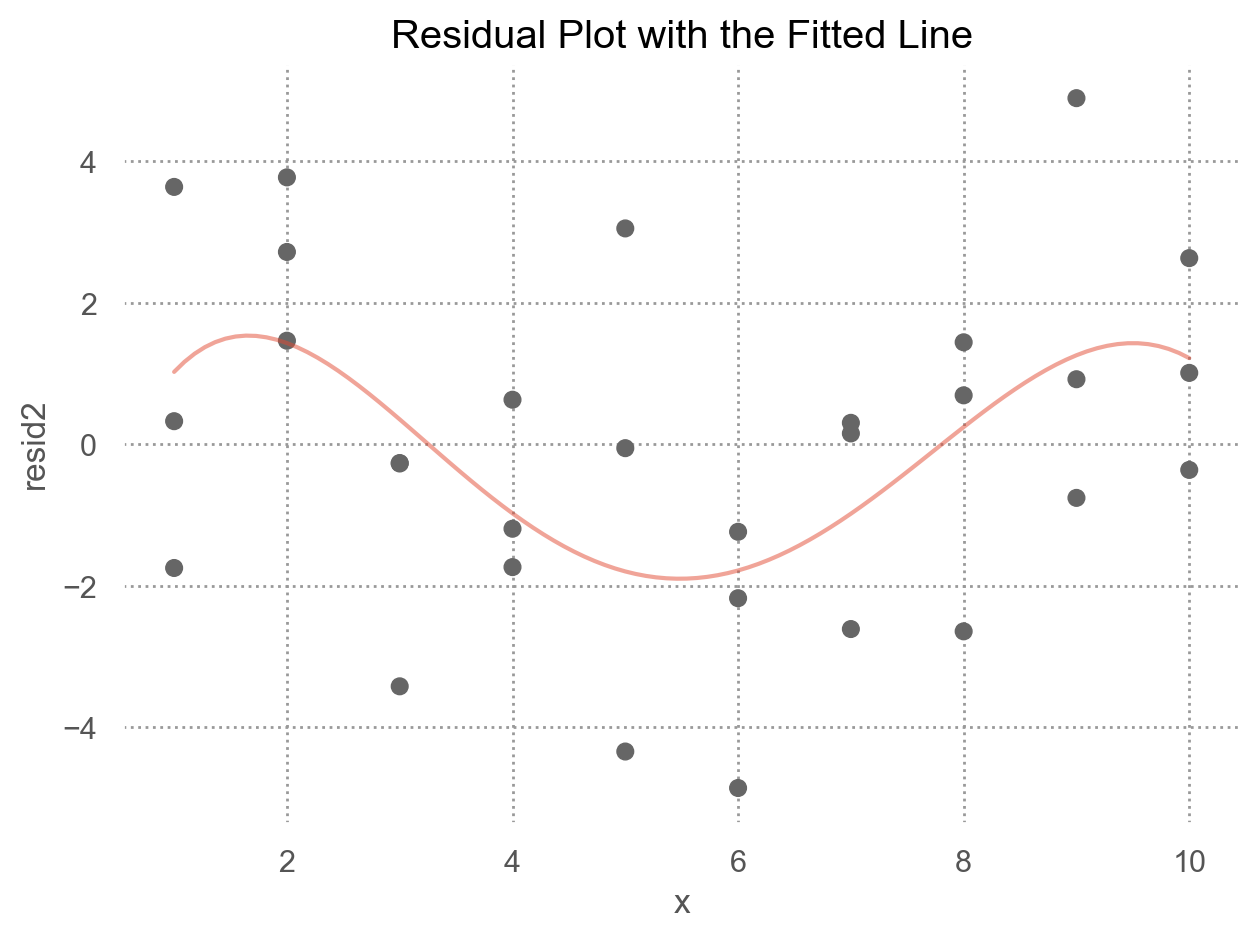
예측 변수와 residuals의 관계를 시각화해서 보면,
(
so.Plot(sim1, x='x', y='resid')
.add(so.Dot())
.add(so.Line(), so.PolyFit(5))
.layout(size=(5, 4))
)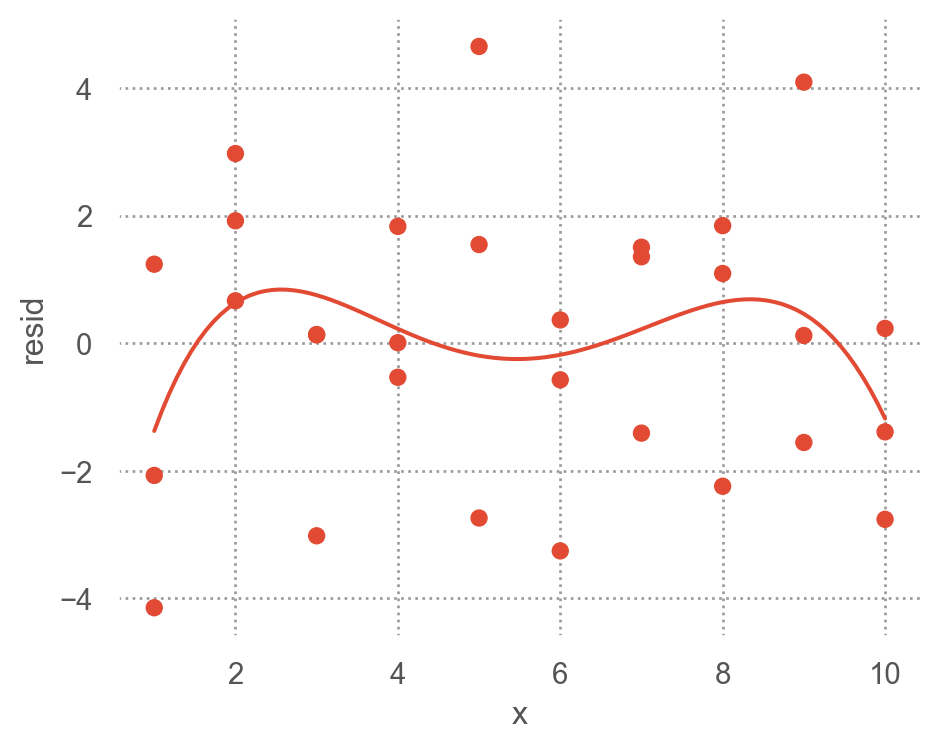
위의 residuals은 특별한 패턴을 보이지 않아야 모델이 데이터의 패턴을 잘 잡아낸 것으로 판단할 수 있음.
아래는 원래 데이터와 일차 선형 모형에 대한 예측값의 관계를 시각화한 것
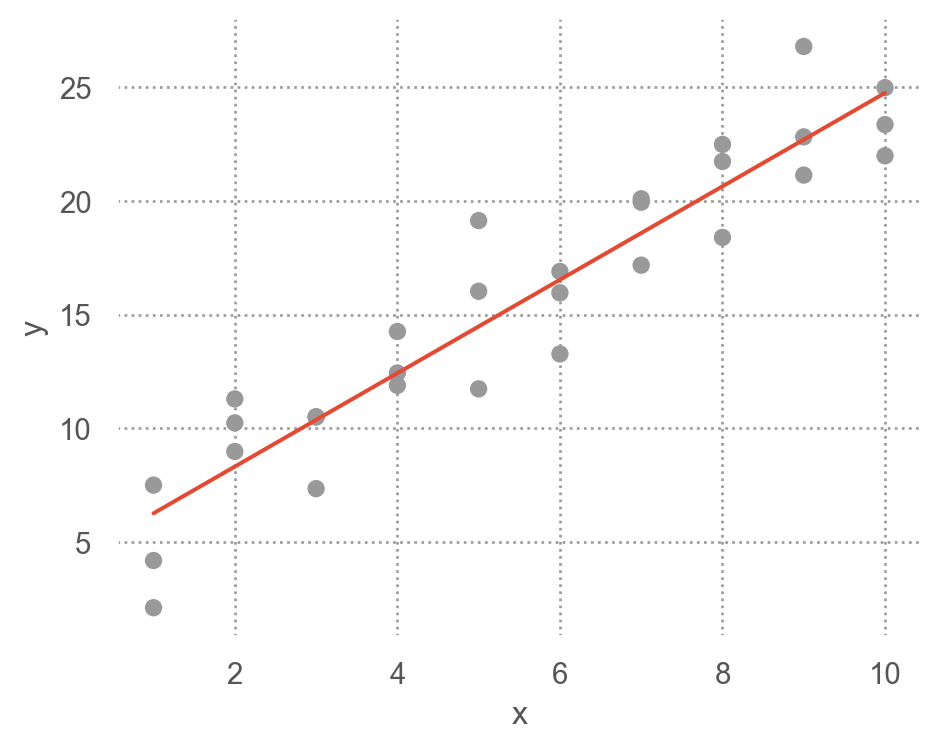
Residuals에 패턴이 보이는 경우
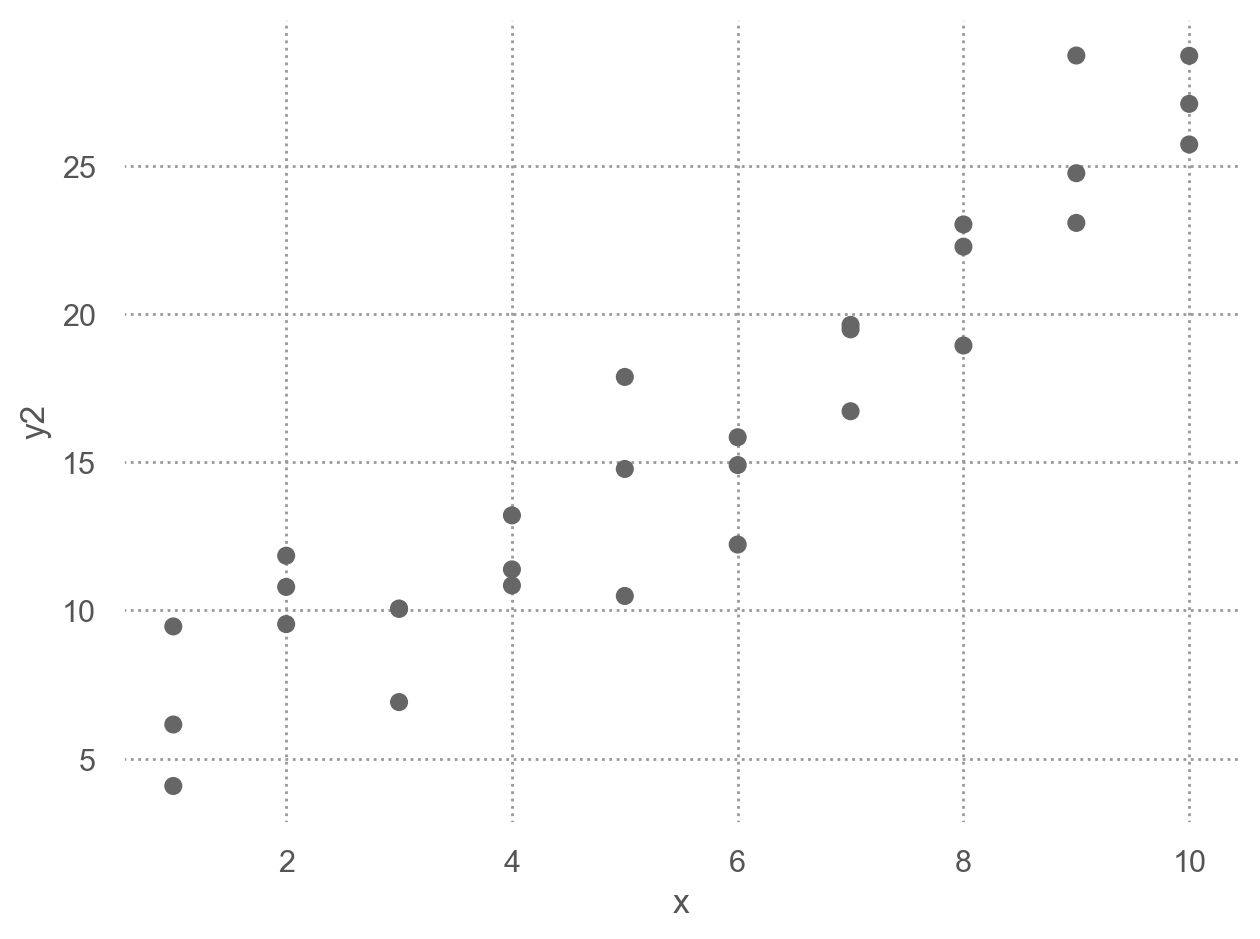
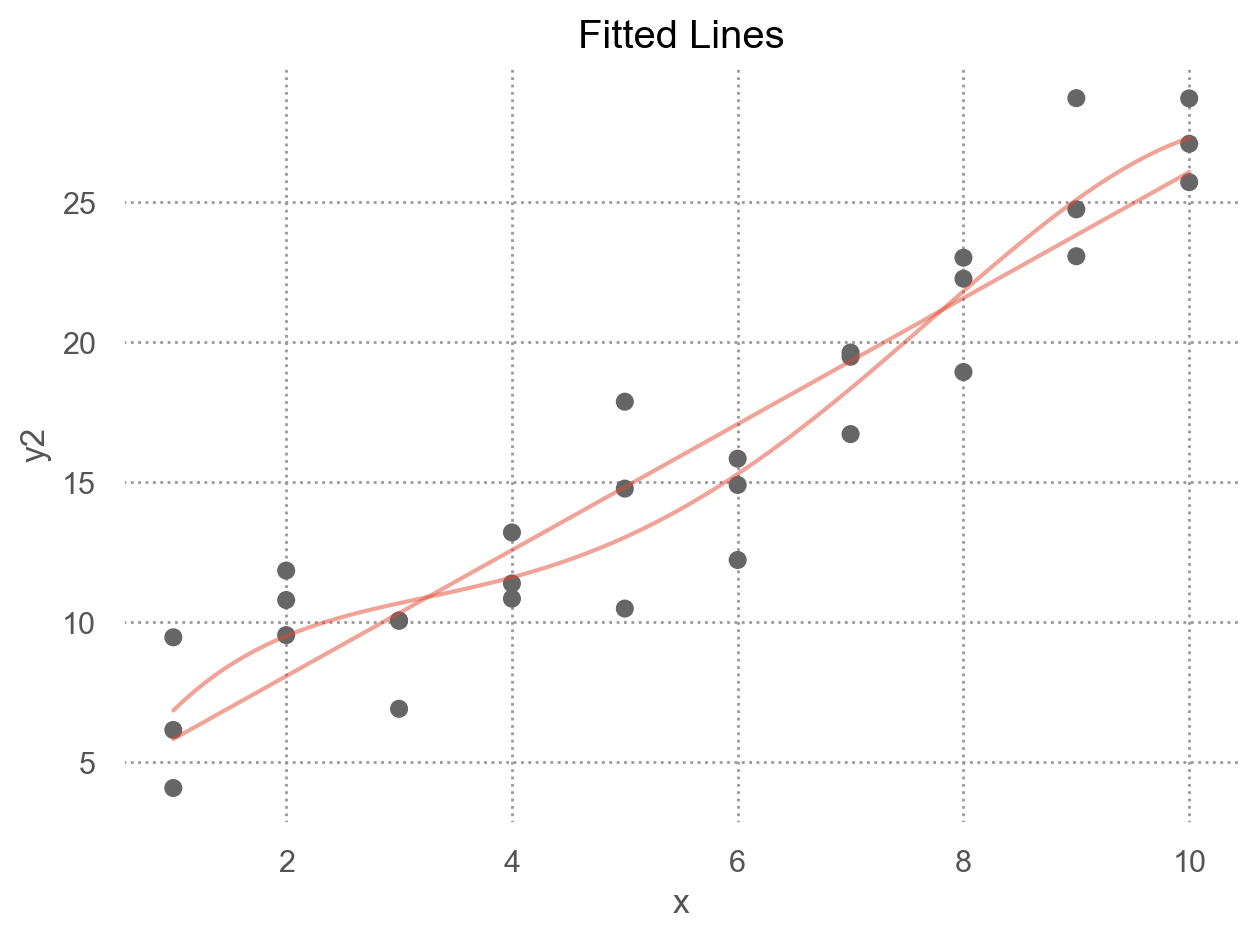
앞서 houses 데이터셋에서 price를 livingArea2와 bedrooms로 예측한 모델의 residuals를 시각화하면,
code
houses["livingArea2"] = houses["livingArea"] / 35.5
mod_houses = ols("price ~ livingArea2 + bedrooms", data=houses).fit()
houses["resid"] = mod_houses.resid
p1 = (
so.Plot(houses, x='livingArea2', y='resid')
.add(so.Dots(color=".5"))
.add(so.Line(), so.PolyFit(5))
)
p2 = (
so.Plot(houses, x='bedrooms', y='resid')
.add(so.Dots(color=".5"))
.add(so.Line(), so.PolyFit(5))
)
display(p1, p2)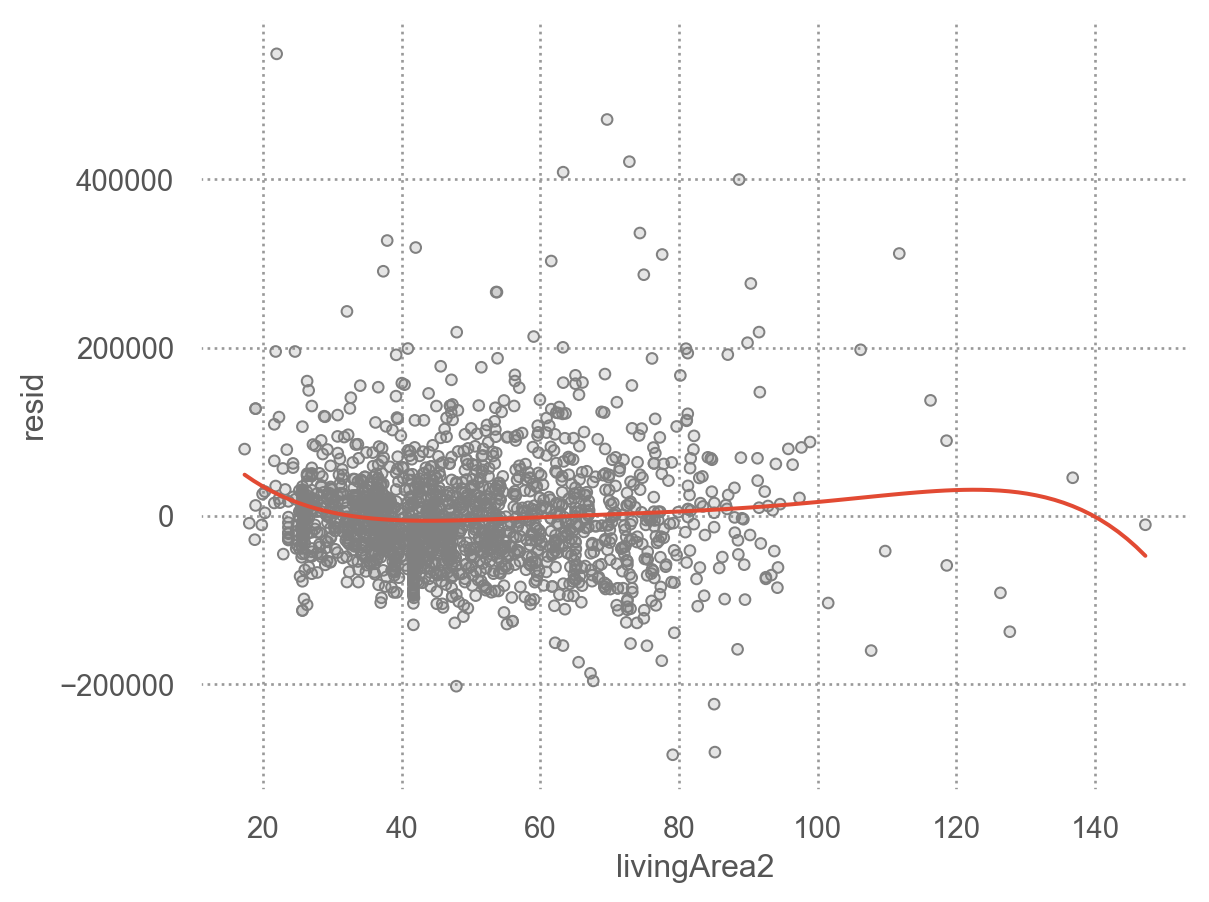
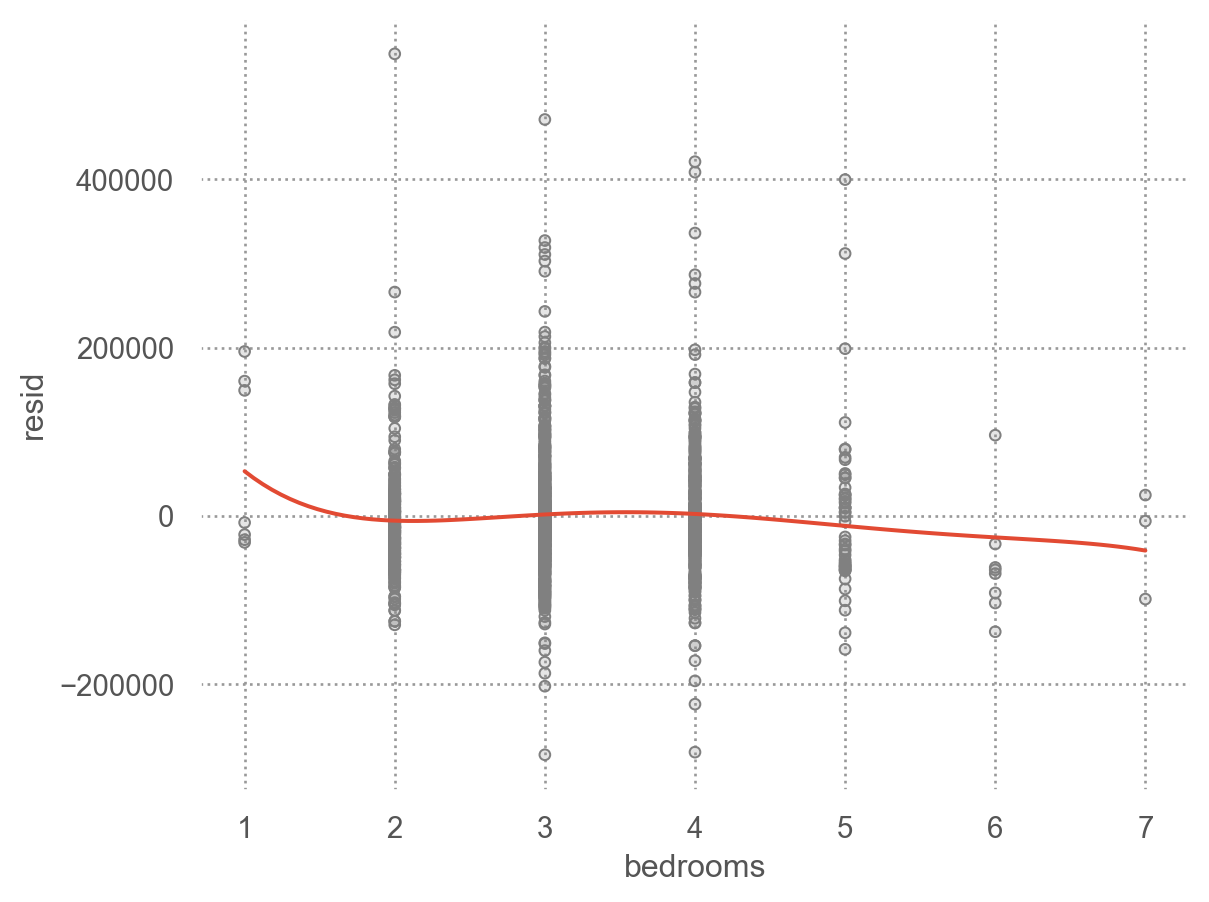
Categorical variables
predictor가 카테고리 변수인 경우 이를 numeric으로 바꾸어야 함
- formula
y ~ sex의 경우, \(y=a_0 +a_1sex\) 로 변환될 수 없음 (성별을 연산할 수 없음) - 실제로, formula는 \(sex[T.male]\) 라는 indicator/dummy variable을 새로 만들어 membership을 나타내 줌: dummy-coding 또는 one-hot encoding 이라고 부름.
- \(y=a_0 +a_1sex[T.male]\) (남성일 때, \(sex[T.male]=1\), 그렇지 않은 경우 0)
- Machine learning 알리고즘 중에 non-parametric 모델은 카테고리 변수를 직접 처리할 수 있음
Patsy의 formula는 편리하게 범주형 변수를 알아서 처리해 주기 때문에 범주형 변수의 복잡한 처리과정을 걱정할 필요가 없음.
직접 변환하려면 pandas의 pd.get_dummies()나 scikit-learn의 OneHotEncoder를 이용
- 2개의 범주인 경우: 한 개의 변수
sex[T.male]이 생성
예를 들어, 다음과 같은 데이터셋이 있을 때, sex로 y를 예측하는 경우
df = pd.DataFrame({"sex": ["male", "female", "female", "male"], "y": [10, 21, 13, 15]})
df sex y
0 male 10
1 female 21
2 female 13
3 male 15# Design matrix
from patsy import dmatrices
y, X = dmatrices('y ~ sex', data=df, return_type="dataframe")
X Intercept sex[T.male]
0 1.00 1.00
1 1.00 0.00
2 1.00 0.00
3 1.00 1.00- 세 개의 범주인 경우: 두 개의 변수
sex[T.male],sex[T.neutral]가 생성 - 일반적으로
n개의 범주를 가진 변수인 경우n-1개의 변수가 생성
df = pd.DataFrame({"sex": ["male", "female", "male", "neutral"], "y": [10, 21, 13, 5]})
df sex y
0 male 10
1 female 21
2 male 13
3 neutral 5y, X = dmatrices("y ~ sex", data=df, return_type="dataframe")
X Intercept sex[T.male] sex[T.neutral]
0 1.00 1.00 0.00
1 1.00 0.00 0.00
2 1.00 1.00 0.00
3 1.00 0.00 1.00직접 변환: pandas의 get_dummies를 이용하거나 scikit-learn의 OneHotEncoder를 이용
# get_dummies() in pandas
pd.get_dummies(df) # drop_first=True, prefix="sex" y sex_female sex_male sex_neutral
0 10 False True False
1 21 True False False
2 13 False True False
3 5 False False True# OneHotEncoder() using scikit-learn
from sklearn.preprocessing import OneHotEncoder
encoder = OneHotEncoder(sparse_output=False) # drop="first"
encoder.fit_transform(df[["sex"]])array([[0., 1., 0.],
[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])실제 예를 들어서 살펴보면,
Data: sim2.csv
sim2 = pd.read_csv("data/sim2.csv")
sim2 x y
0 a 1.94
1 a 1.18
2 a 1.24
.. .. ...
37 d 2.13
38 d 2.49
39 d 0.30
[40 rows x 2 columns]sim2.plot.scatter(x="x", y="y")
plt.show()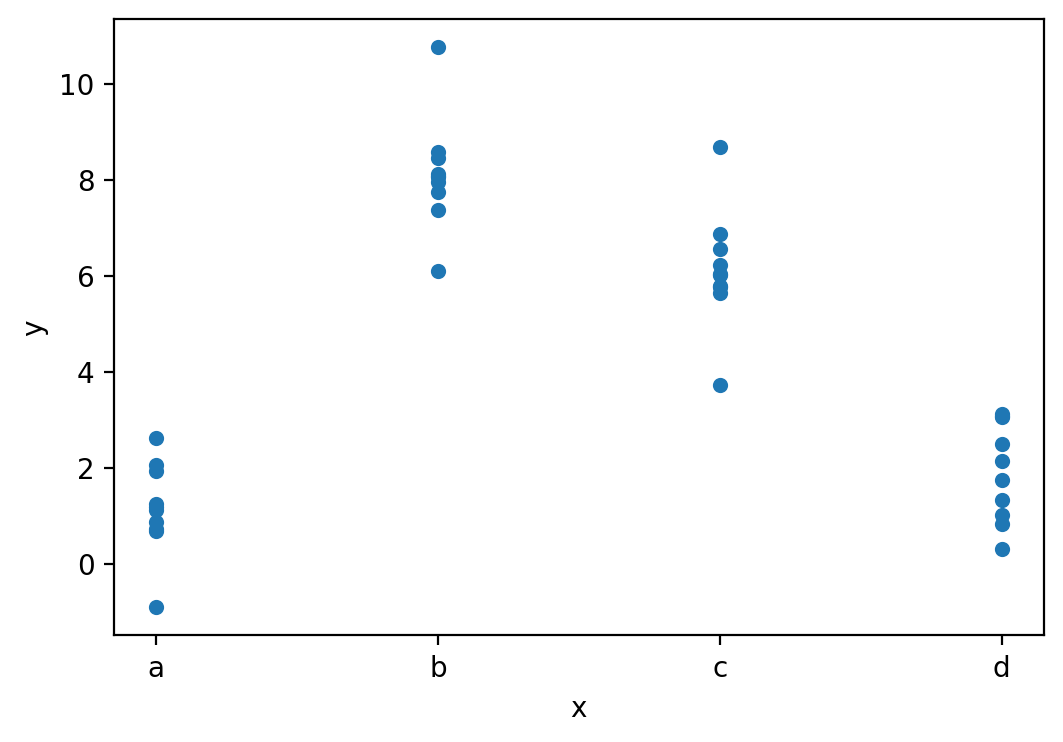
Fit a model to Data
mod2 = ols('y ~ x', data=sim2).fit()
mod2.paramsIntercept 1.15
x[T.b] 6.96
x[T.c] 4.98
x[T.d] 0.76
dtype: float64모델을 이용해 새로운 데이터의 예측값을 구하면
grid = pd.DataFrame({"x": list("abcd")}) # new data
grid["pred"] = mod2.predict(grid)
grid x pred
0 a 1.15
1 b 8.12
2 c 6.13
3 d 1.91예측값을 시각화하면,
각 카테고리 별로 평균값으로 예측… why?
# plt.scatter scatter plot, 점의 내부가 채워지지 않도록 설정
plt.scatter(sim2["x"], sim2["y"])
plt.scatter(grid["x"], grid["pred"], color="red")
plt.show()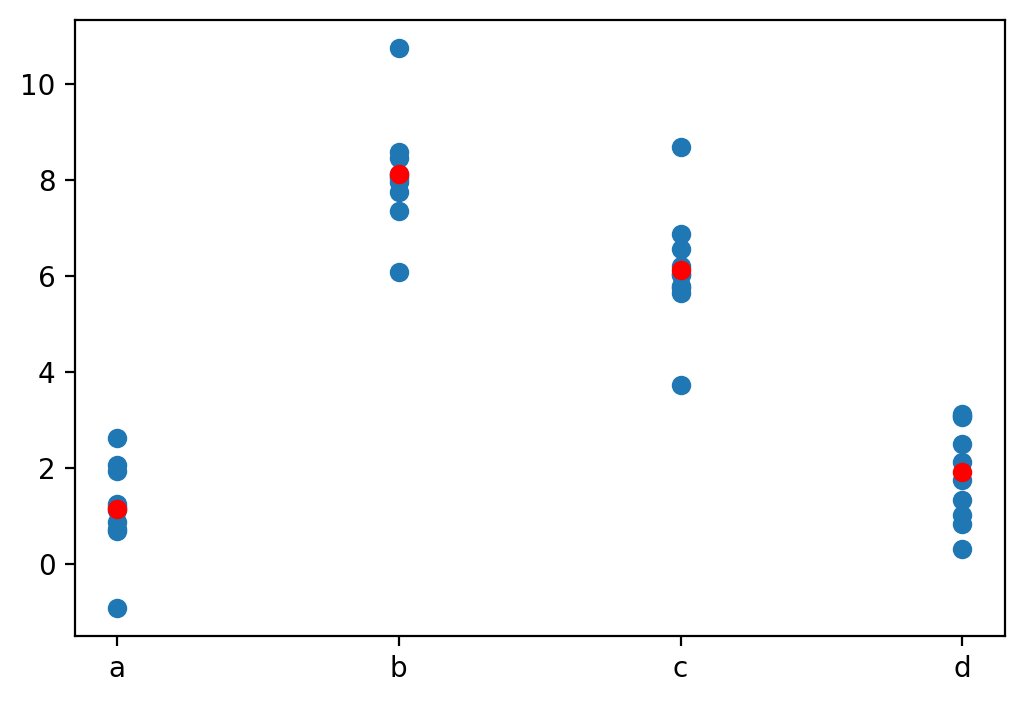
fitted model의 파라미터를 보면,
formula y ~ x는 실제 \(\hat{y} = a_0 + a_1x[T.b] + a_2 x[T.c] + a_3 x[T.d]\) 으로 해석
y, X = dmatrices("y ~ x", data=sim2, return_type="dataframe")
pd.concat([X, sim2["x"]], axis=1).sample(8) Intercept x[T.b] x[T.c] x[T.d] x
25 1.00 0.00 1.00 0.00 c
37 1.00 0.00 0.00 1.00 d
32 1.00 0.00 0.00 1.00 d
.. ... ... ... ... ..
10 1.00 1.00 0.00 0.00 b
1 1.00 0.00 0.00 0.00 a
2 1.00 0.00 0.00 0.00 a
[8 rows x 5 columns]SaratogaHouses 데이터셋에 적용해 보면,
houses = sm.datasets.get_rdataset("SaratogaHouses", "mosaicData").data
sns.boxplot(y="price", x="fuel", data=houses, fill=False)
plt.show()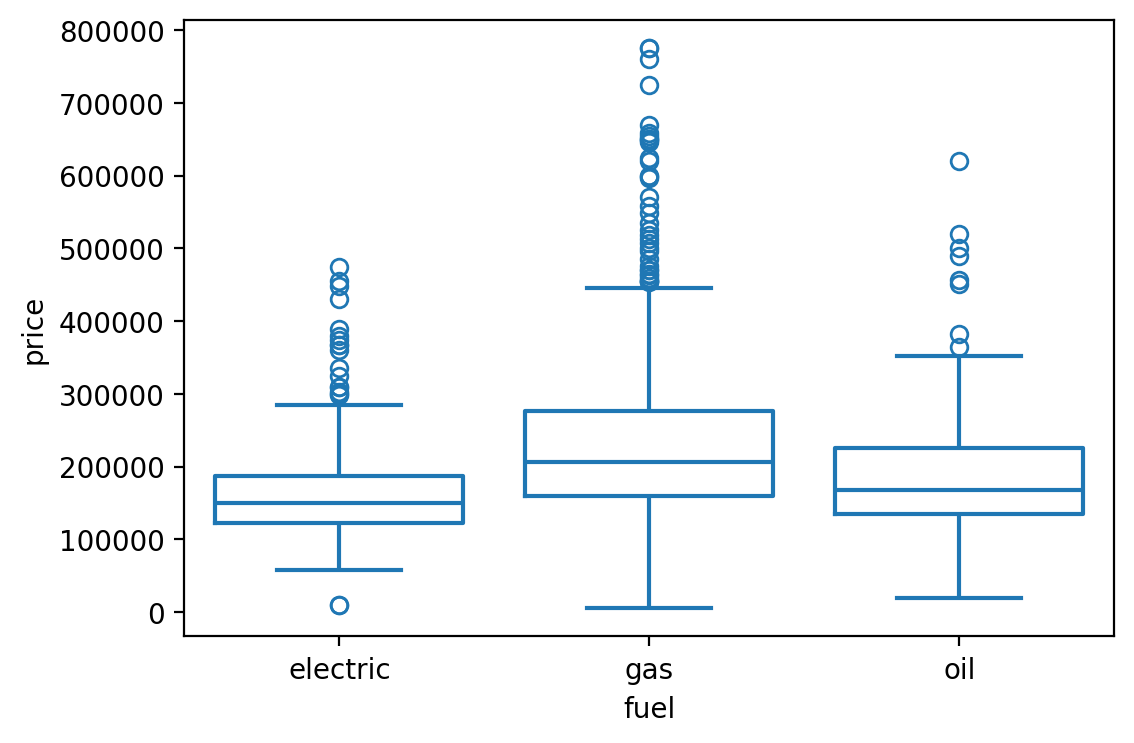
mod_houses = ols("price ~ fuel", data=houses)
mod_houses.fit().paramsIntercept 164937.57
fuel[T.gas] 63597.52
fuel[T.oil] 23796.83
dtype: float64\(\widehat{price}\) = $164,937 + $635,979· fuel[T.gas] + $23,796·fuel[T.oil]
- 절편 $164,937: dummy variables의 값이 모두 0일 때, 즉 electric일 때의 평균 가격
fuel[T.gas]= 0,fuel[T.oil]= 0,fuel[T.electric]= 1 일 때- 각 기울기는 electric일과 해당 fuel 종류의 평균 가격의 차이를 의미
y, X = dmatrices("price ~ fuel", data=houses, return_type="dataframe")
X Intercept fuel[T.gas] fuel[T.oil]
0 1.00 0.00 0.00
1 1.00 1.00 0.00
2 1.00 1.00 0.00
... ... ... ...
1725 1.00 1.00 0.00
1726 1.00 1.00 0.00
1727 1.00 1.00 0.00
[1728 rows x 3 columns]Interactions
Two continuous
두 연속변수가 서로 상호작용하는 경우: not additive, but multiplicative
- 각각의 효과가 더해지는 것을 넘어서서 서로의 효과를 증폭시키거나 감소시키는 경우
- 강수량과 풍속이 함께 항공편의 지연을 가중시키는 경우
- 운동량과 식사량이 함께 체중 감량에 영향을 미치는 경우
Show the code
np.random.seed(123)
x1 = np.random.uniform(0, 10, 200)
x2 = 2*x1 - 1 + np.random.normal(0, 12, 200)
y = x1 + x2 + x1*x2 + np.random.normal(0, 50, 200)
df = pd.DataFrame(dict(precip=x1, wind=x2, delay=y))
df precip wind delay
0 6.96 4.04 95.70
1 2.86 5.60 31.23
2 2.27 8.37 -30.97
.. ... ... ...
197 7.45 16.38 186.67
198 4.73 -18.56 -96.90
199 1.22 -5.63 -22.95
[200 rows x 3 columns]# additive model
mod1 = ols('delay ~ precip + wind', data=df).fit()
# interaction model
mod2 = ols('delay ~ precip + wind + precip:wind', data=df).fit()mod2: y ~ x1 + x2 + x1:x2는 \(\hat{y} = a_0 + a_1x_1 + a_2x_2 + a_3x_1x_2\) 로 변환되고,
변형하면, \(\hat{y} = a_0 + a_1x_1 + (a_2 + a_3x_1)x_2\)
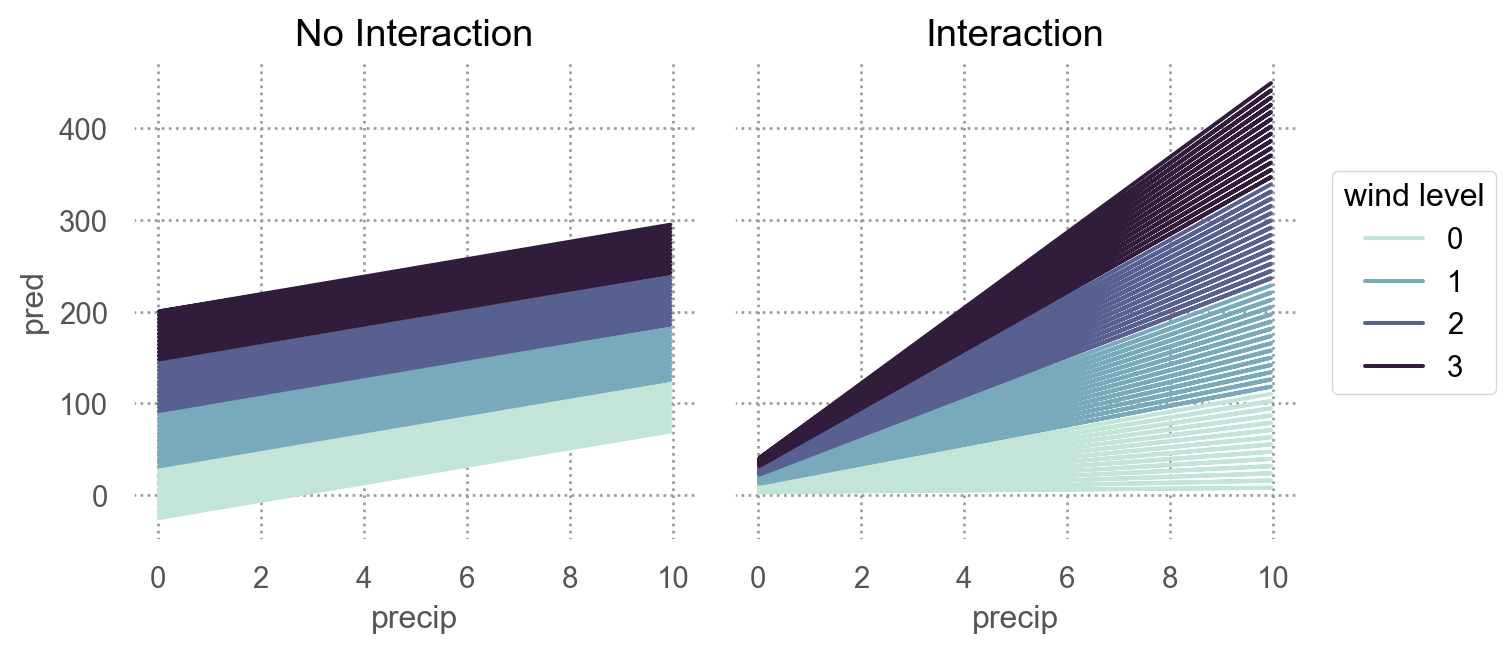
Continuous and Categorical
연속변수와 범주형 변수가 서로 상호작용하는 경우
- 운동량이 건강에 미치는 효과: 혼자 vs. 단체
Data: sim3.csv
sim3 = pd.read_csv("data/sim3.csv")
sim3 x1 x2 rep y sd
0 1 a 1 -0.57 2
1 1 a 2 1.18 2
2 1 a 3 2.24 2
.. .. .. ... ... ..
117 10 d 1 6.56 2
118 10 d 2 5.06 2
119 10 d 3 5.14 2
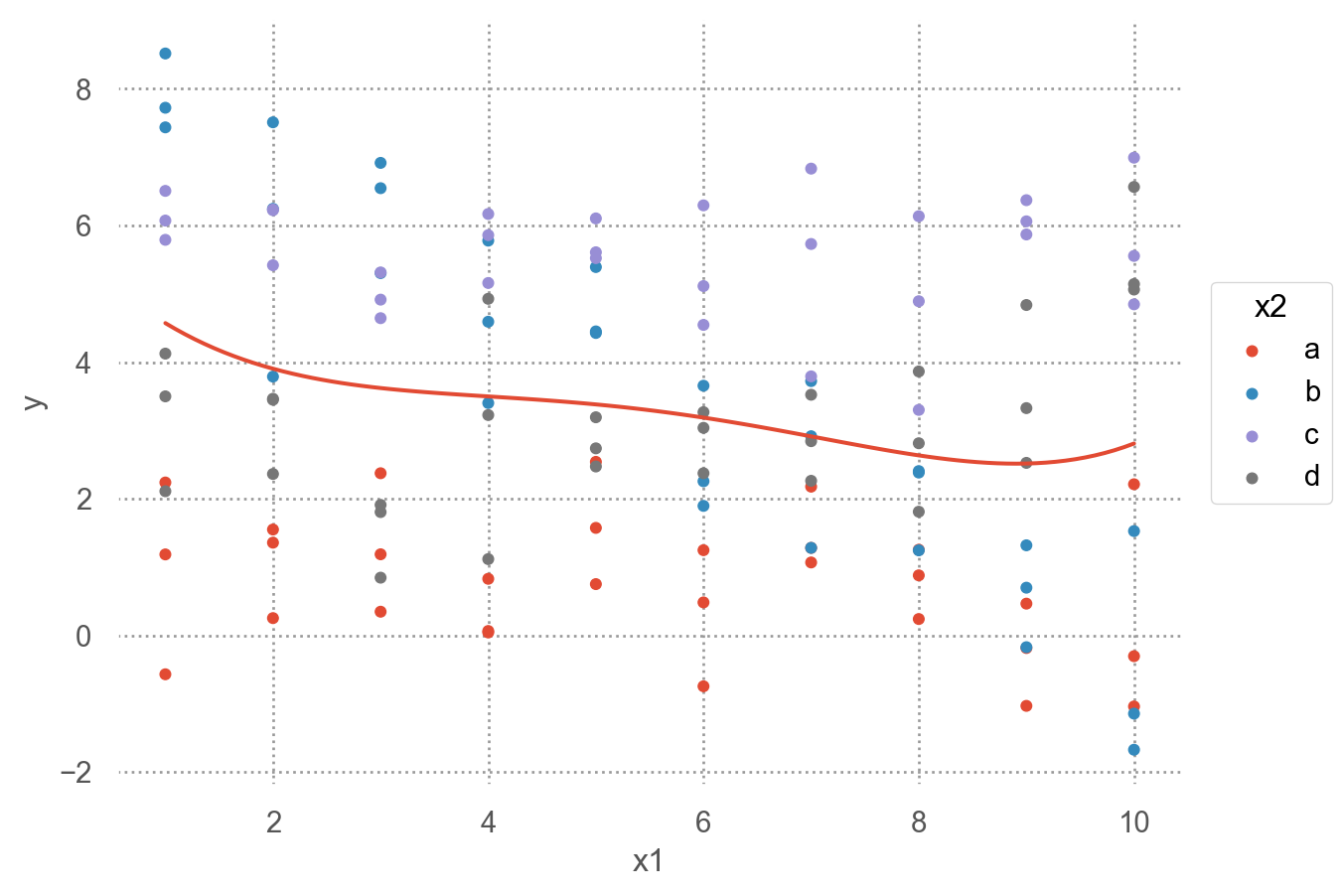
[120 rows x 5 columns]Code
(
so.Plot(sim3, x='x1', y='y', color='x2')
.add(so.Dot(pointsize=4))
.add(so.Line(), so.PolyFit(5), color=None)
)
두 가지 모델로 fit할 수 있음
mod1 = ols('y ~ x1 + x2', data=sim3).fit()
mod2 = ols('y ~ x1 * x2', data=sim3).fit() # 같은 의미 'y ~ x1 + x2 + x1:x2'formula y ~ x1 * x2는 \(\hat{y} = a_0 + a_1x_1 + a_2x_2 + a_3x_1x_2\)로 변환됨
하지만, 여기서는 x2가 범주형 변수라 dummy-coding후 적용됨.
Design matrix를 확인해 보면,
y, X = dmatrices("y ~ x1 + x2", data=sim3, return_type="dataframe")
X.iloc[:, 1:] x2[T.b] x2[T.c] x2[T.d] x1
0 0.00 0.00 0.00 1.00
1 0.00 0.00 0.00 1.00
2 0.00 0.00 0.00 1.00
.. ... ... ... ...
117 0.00 0.00 1.00 10.00
118 0.00 0.00 1.00 10.00
119 0.00 0.00 1.00 10.00
[120 rows x 4 columns]y, X = dmatrices("y ~ x1 * x2", data=sim3, return_type="dataframe")
X.iloc[:, 1:] x2[T.b] x2[T.c] x2[T.d] x1 x1:x2[T.b] x1:x2[T.c] x1:x2[T.d]
0 0.00 0.00 0.00 1.00 0.00 0.00 0.00
1 0.00 0.00 0.00 1.00 0.00 0.00 0.00
2 0.00 0.00 0.00 1.00 0.00 0.00 0.00
.. ... ... ... ... ... ... ...
117 0.00 0.00 1.00 10.00 0.00 0.00 10.00
118 0.00 0.00 1.00 10.00 0.00 0.00 10.00
119 0.00 0.00 1.00 10.00 0.00 0.00 10.00
[120 rows x 7 columns]grid = sim3.value_counts(["x1", "x2"]).reset_index().drop(columns="count")
grid["mod1"] = mod1.predict(grid)
grid["mod2"] = mod2.predict(grid)
grid_long = grid.melt(id_vars=["x1", "x2"], var_name="model", value_name="pred")
grid_full = grid_long.merge(sim3[["x1", "x2", "y"]])grid_full x1 x2 model pred y
0 1 a mod1 1.67 -0.57
1 1 a mod1 1.67 1.18
2 1 a mod1 1.67 2.24
.. .. .. ... ... ...
237 10 d mod2 3.98 6.56
238 10 d mod2 3.98 5.06
239 10 d mod2 3.98 5.14
[240 rows x 5 columns]Code
(
so.Plot(grid_full, x="x1", y="y", color="x2")
.add(so.Dot(pointsize=4))
.add(so.Line(), y="pred")
.facet("model")
.layout(size=(8, 5))
)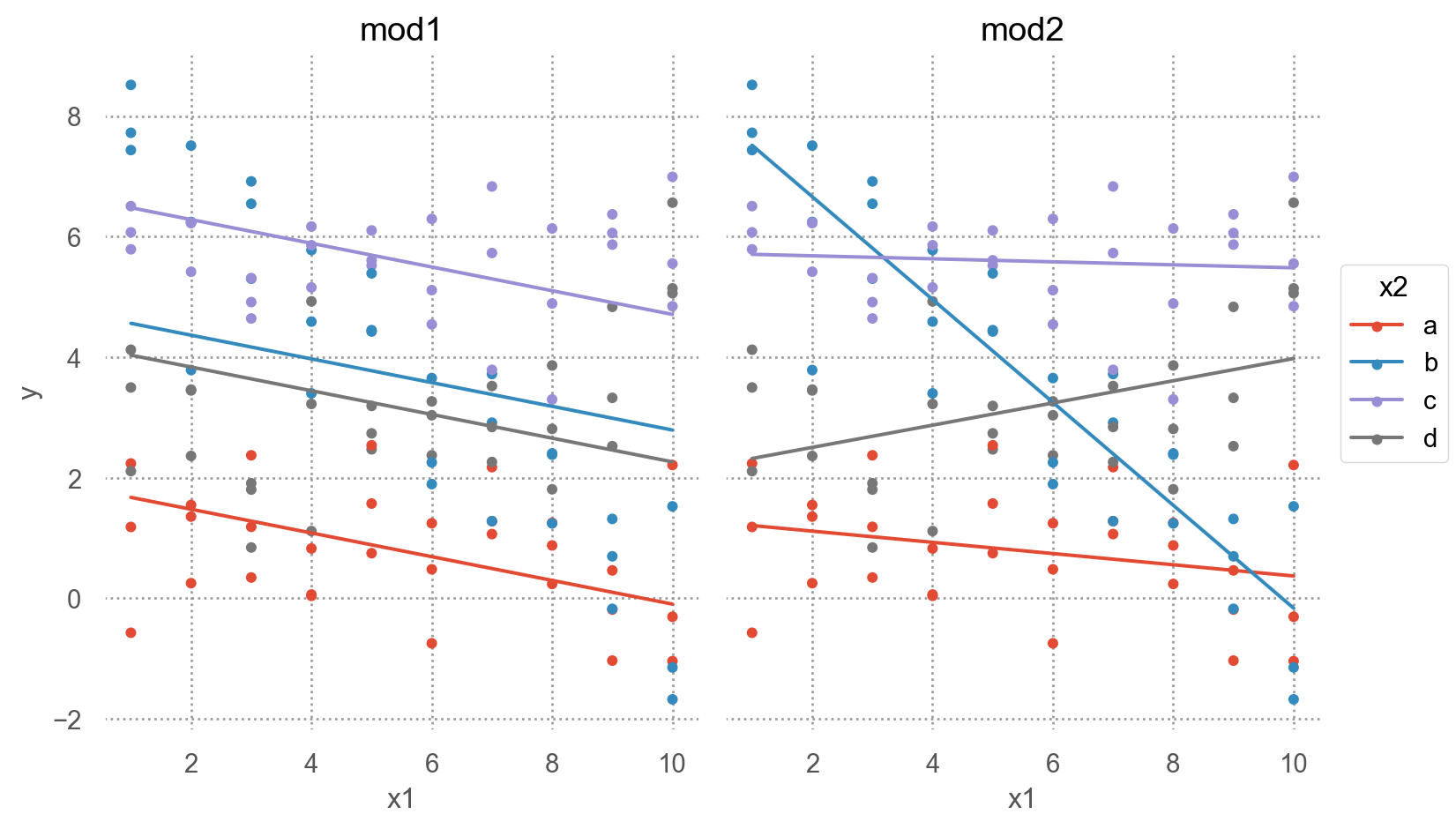
- interaction이 없는 모형 mod1의 경우, 네 범주에 대해 기울기가 동일하고 절편의 차이만 존재
- interaction이 있는 모형 mod2의 경우, 네 범주에 대해 기울기가 다르고 절편도 다름
\(y = a_0 + a_1x_1 + a_2x_2 + a_3x_1x_2\)에서 \(x_1x_2\)항이 기울기를 변할 수 있도록 해줌 \(y = a_0 + a_2x_2 + (a_1 + a_3x_2)x_1\)으로 변형하면, \(x_1\)의 기울기는 \(a_1 + a_3 x_2\)
mod1 = ols('y ~ x1 + x2', data=sim3).fit()
mod1.params
# Intercept 1.87
# x2[T.b] 2.89
# x2[T.c] 4.81
# x2[T.d] 2.36
# x1 -0.20
mod2 = ols('y ~ x1 * x2', data=sim3).fit() # 같은 의미 'y ~ x1 + x2 + x1:x2'
mod2.params
# Intercept 1.30
# x2[T.b] 7.07
# x2[T.c] 4.43
# x2[T.d] 0.83
# x1 -0.09
# x1:x2[T.b] -0.76
# x1:x2[T.c] 0.07
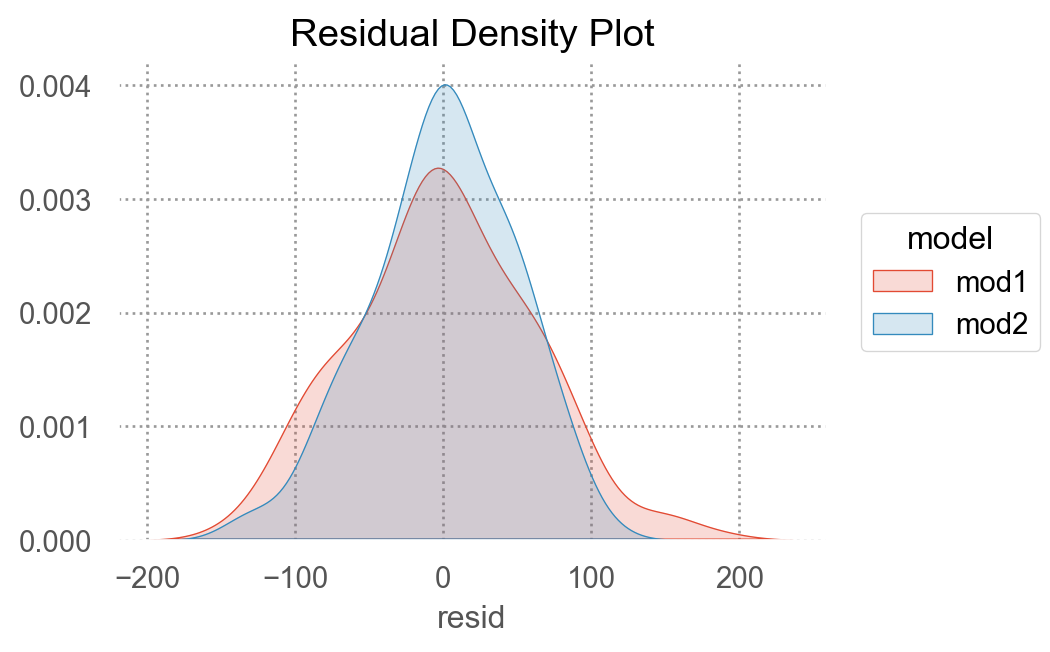
# x1:x2[T.d] 0.28두 모형을 비교하여 중 더 나은 모형을 선택하기 위해, residuals을 차이를 살펴보면,
sim3["mod1"] = mod1.resid
sim3["mod2"] = mod2.resid
sim3_long = sim3.melt(
id_vars=["x1", "x2"],
value_vars=["mod1", "mod2"],
var_name="model",
value_name="resid",
)
sim3_long x1 x2 model resid
0 1 a mod1 -2.25
1 1 a mod1 -0.49
2 1 a mod1 0.56
3 1 b mod1 2.87
.. .. .. ... ...
236 10 c mod2 -0.64
237 10 d mod2 2.59
238 10 d mod2 1.08
239 10 d mod2 1.16
[240 rows x 4 columns]Code
(
so.Plot(sim3_long, x="x1", y="resid", color="x2")
.add(so.Dot(pointsize=4))
.add(so.Line(linestyle=":", color=".5"), so.Agg(lambda x: 0))
.facet("x2", "model")
.layout(size=(9, 6))
.scale(color="Set2")
)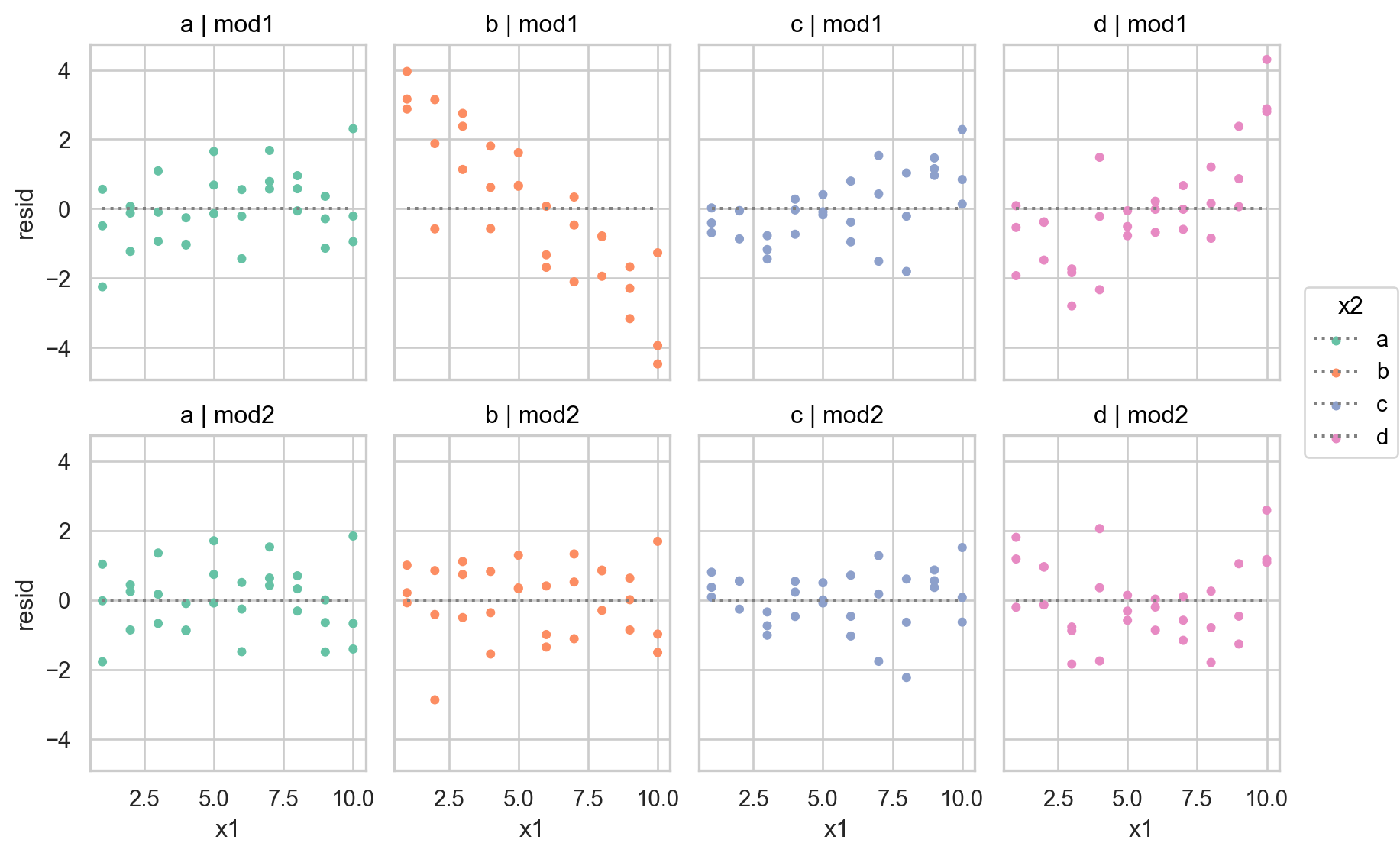
- 둘 중 어떤 모델이 더 나은지에 대한 정확한 통계적 비교가 가능하나 (잔차의 제곱의 평균인 RMSE나 잔차의 절대값의 평균인 MAE 등)
- 여기서는 직관적으로 어느 모델이 데이터의 패턴을 더 잘 잡아냈는지를 평가하는 것으로 충분
- 잔차를 직접 들여다봄으로써, 어느 부분에서 어떻게 예측이 잘 되었는지, 잘 안 되었는지를 면밀히 검사할 수 있음
- interaction 항이 있는 모형이 더 나은 모형
SaratogaHouses 데이터에서 가령, livingArea와 centralAir의 interaction을 살펴보면,
houses = sm.datasets.get_rdataset("SaratogaHouses", "mosaicData").data
(
so.Plot(houses, x='livingArea', y='price')
.add(so.Dots(color='.6'))
.add(so.Line(color="orangered"), so.PolyFit(1))
.facet("centralAir")
.label(title="Central Air: {}".format)
.layout(size=(8, 4))
)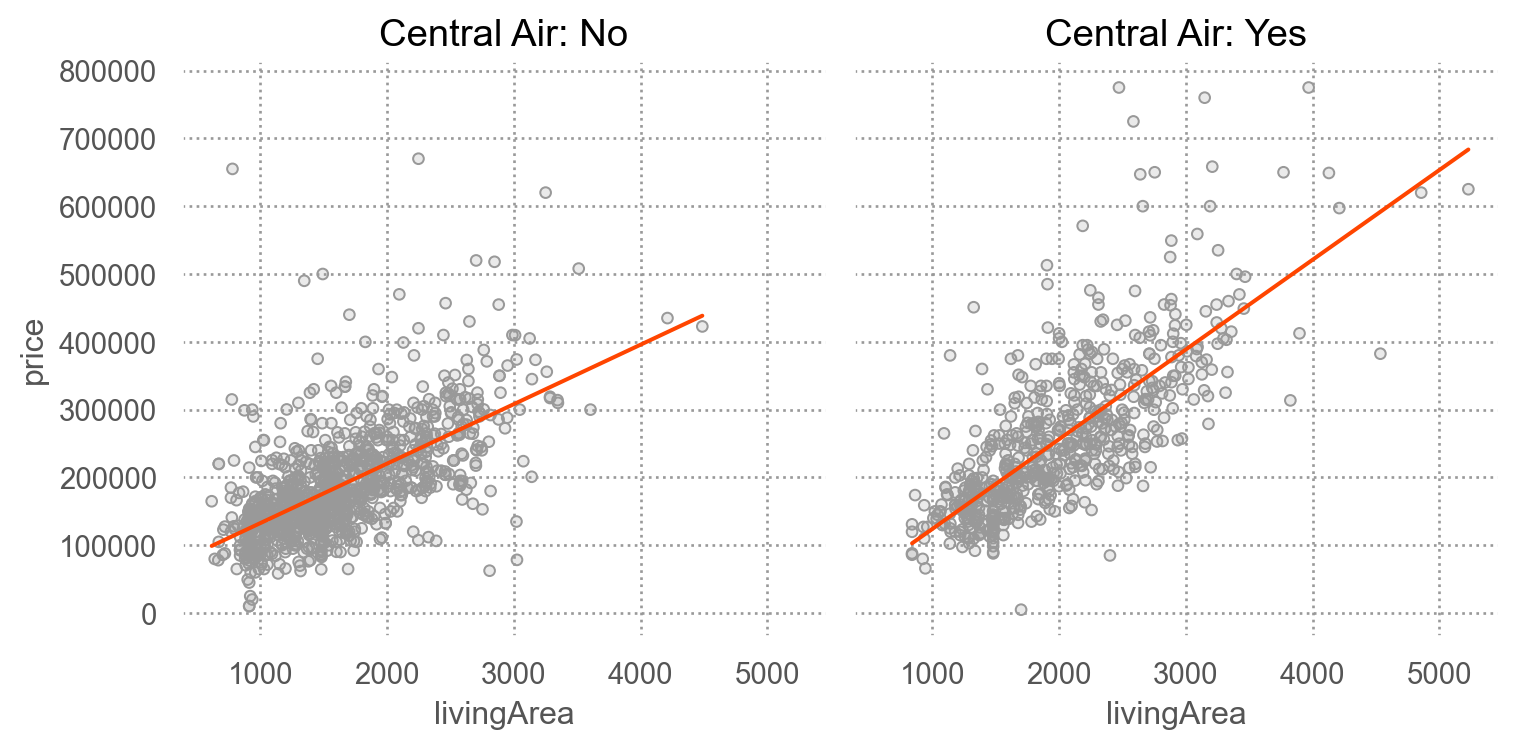
mod1 = ols('price ~ livingArea + centralAir', data=houses).fit()
mod2 = ols('price ~ livingArea * centralAir', data=houses).fit()
display(mod1.params, mod2.params)Intercept 14144.05
centralAir[T.Yes] 28450.58
livingArea 106.76
dtype: float64Intercept 44977.64
centralAir[T.Yes] -53225.75
livingArea 87.72
livingArea:centralAir[T.Yes] 44.61
dtype: float64R-squared 비교
display(mod1.rsquared, mod2.rsquared)0.52532231493391370.5430362101820772